https://arxiv.org/abs/2409.08022
De novo design of high-affinity protein binders with AlphaProteo
Computational design of protein-binding proteins is a fundamental capability with broad utility in biomedical research and biotechnology. Recent methods have made strides against some target proteins, but on-demand creation of high-affinity binders without
arxiv.org
초록
단백질-결합 단백질의 컴퓨터 설계는 생의학 연구와 생명공학에서 폭넓은 유용성을 가진 근본적인 능력입니다. 최근의 방법들은 몇몇 표적 단백질에 대해 성과를 이루었지만, 실험적인 테스트를 여러 번 반복하지 않고도 높은 친화력의 결합체를 즉시 만드는 것은 여전히 해결되지 않은 과제입니다. 이 기술 보고서에서는 단백질 설계를 위한 기계 학습 모델의 집합인 AlphaProteo를 소개하고, de novo 결합체 설계 문제에서의 성능을 자세히 설명합니다. AlphaProteo를 통해 우리는 7개의 표적 단백질에 대해 기존 최고 방법들보다 3배에서 300배 더 나은 결합 친화도와 더 높은 실험적 성공률을 달성했습니다. 우리의 결과는 AlphaProteo가 "즉시 사용 가능한" 결합체를 중간 처리량의 스크리닝 한 번만으로도 추가 최적화 없이 다수의 연구 응용에서 생성할 수 있음을 시사합니다.
실험적 주요 결과
- AlphaProteo 단백질 설계 시스템을 소개하고, 8개의 구조적으로 다양한 표적 단백질에 대해 설계된 결합체를 실험적으로 테스트하였습니다.
- 7개의 표적 중 9%에서 88%의 디자인이 실험실에서 성공적인 결합체로 실험적으로 확인되었습니다. 이 수치는 기존의 최고 방법보다 높으며, 다른 방법보다 5배에서 100배 높은 수치입니다. 이들 중 하나에 대해서는 처음으로 컴퓨터 설계된 결합체를 보고합니다.
- 수백 개의 PDB 표적 단백질에 대한 AlphaProteo의 in silico 성능은 이 7개의 표적과 유사하며, 이는 이 방법이 널리 일반화될 가능성이 있음을 시사합니다. 우리는 PDB 스크리닝 중 가장 어려운 표적 중 하나를 8번째 표적으로 선택했지만 결합체를 얻는 데 실패했습니다.
- 우리는 네 개의 표적에 대해 80-960 피코몰라 친화도, 다른 세 개의 표적에 대해 낮은 나노몰라 친화도를 가지는 결합체를, 고처리량 스크리닝이나 실험적 친화도 최적화 없이 얻었습니다. 7개의 표적에 대해 우리의 디자인은 기존의 최상 설계된 결합체보다 3배에서 300배 더 나은 결합 친화도를 가집니다.
- 두 표적에 대해 생물학적 기능을 테스트한 결과, 인간 세포에서 VEGF 신호 억제 및 Vero 원숭이 세포에서 SARS-CoV-2 중화를 입증했습니다.
- Cryo-EM 및 X선 결정학은 설계된 결합체와 결합체-표적 복합체의 구조를 확인했습니다.
-----
- PDB 표적 단백질에 대한 AlphaProteo의 성능:
- AlphaProteo라는 모델을 사용해서 실험 전에 컴퓨터로 수백 개의 단백질에 대해 테스트를 진행했어요. 이 성능이 실제 실험에서 테스트한 7개의 단백질에 대한 성능과 비슷하게 나왔다는 거죠. 이는 AlphaProteo가 다양한 단백질에 대해 효과적으로 일반화될 수 있다는 것을 의미해요. 즉, 특정 단백질만 잘 맞는 것이 아니라, 여러 종류의 단백질에 대해서도 성능이 좋을 가능성이 있다는 의미입니다.
- 하지만 가장 어려운 표적 단백질 중 하나를 테스트한 결과, 이 경우에는 결합체(바인더)를 성공적으로 얻지 못했어요. 즉, 모든 단백질에 대해 완벽하게 동작하는 것은 아직 아니라는 것이죠.
- 결합 친화도:
- AlphaProteo는 네 개의 표적 단백질에 대해 매우 높은 친화도(결합 강도)를 가진 결합체를 만들어냈습니다. 이 친화도는 80-960 피코몰라(pM) 수준으로, 매우 강하게 결합한다는 뜻이에요.
- 또 다른 세 개의 표적에 대해서는 낮은 나노몰라(nM) 수준의 친화도를 가졌어요. 이 역시 강한 결합을 의미하는데, 피코몰라보다는 살짝 약해요.
- 중요한 점은, 이런 성과를 내는 데 있어서 굉장히 대규모 실험(고처리량 스크리닝)이나 추가적인 실험적 최적화 없이도 이뤄냈다는 것입니다. 그리고 이 성과는 기존의 가장 잘 설계된 결합체보다 3배에서 300배 더 좋은 결합 성능을 보였다는 것입니다.
- 생물학적 기능 테스트:
- AlphaProteo로 설계된 두 개의 결합체는 실제로 생물학적 기능을 하는지 테스트되었어요.
- 첫 번째로, 인간 세포에서 VEGF 신호를 억제하는 것을 확인했습니다. VEGF는 혈관 형성에 중요한 역할을 하는 단백질인데, 억제하면 혈관 형성을 막을 수 있어서 항암 연구 등에서 중요한 역할을 할 수 있어요.
- 두 번째로, SARS-CoV-2(코로나 바이러스)를 Vero 원숭이 세포에서 중화하는 것도 확인되었어요. 이는 바이러스의 감염을 막는 데 도움이 될 수 있다는 것을 의미합니다.
- AlphaProteo로 설계된 두 개의 결합체는 실제로 생물학적 기능을 하는지 테스트되었어요.
쉽게 말해, AlphaProteo라는 모델이 기존의 설계 방법보다 훨씬 강력한 결합체를 효율적으로 설계해내며, 그 결합체가 생물학적으로도 실제로 유용하다는 것을 입증한 내용입니다.
-----
목차
- 서론
- 결과 2.1 중간 처리량 스크리닝을 통한 서브나노몰라 친화도 결합체 2.1.1 표적당 96웰 플레이트 하나에 여러 결합 히트 2.1.2 7개의 표적에 대한 최첨단 결합 친화도 2.1.3 설계된 결합체가 표적 에피토프에 결합함 2.1.4 디자인이 특정 결합을 가지며 구조적으로 다양함 2.2 결합체의 기능 및 구조적 검증 2.2.1 라이브 바이러스 중화 실험에서 SARS-CoV-2 변이체 중화 2.2.2 세포에서 VEGF 수용체 하위 신호 억제 2.2.3 결합체-표적 복합체의 실험적 구조가 결합 모드와 구조를 확인
- 결론
부록 정보 S1. 실험 방법 S1.1 표적 단백질의 발현 및 정제 S1.2 효모 표면 디스플레이 및 유세포 분석 S1.2.1 초기 결합 스크리닝 S1.2.2 인터페이스 돌연변이, 경쟁 억제, 특이성 실험 S1.3 설계된 결합체의 발현 및 정제 S1.4 결합 친화도 / 결합 해리 상수(K_D)의 측정 S1.4.1 동질 시간 분해 형광(HTRF) S1.4.2 바이오-레이어 간섭계(BLI) S1.5 원형 이색성(CD) 분광법 S1.6 HUVEC에서 VEGF-A 신호의 웨스턴 블롯 분석 S1.7 SARS-CoV-2 바이러스 중화 실험 S1.8 Cryo-EM 샘플 준비, 데이터 수집 및 이미지 처리 S1.9 X선 결정학 샘플 준비, 데이터 처리 및 구조 해석 S2 AlphaProteo의 반복적 개발 및 in silico 벤치마킹 S2.1 AF2 기반 벤치마크 S2.2 AF3 기반 벤치마크 S3 PDB 표적의 in silico 스크리닝 S4 다른 설계 방법과의 비교 S4.1 RFdiffusion과의 실험적 성공률 비교 S4.2 다른 방법들과의 결합 친화도(K_D) 비교
부록 그림 부록 표 부록 참고 문헌
1. 서론
단백질-단백질 상호작용은 단백질 기능의 근본적인 측면이며, 단백질-결합 단백질은 치료제, 진단제 및 생의학 연구의 기본적인 구성 요소입니다 [19, 29]. 전통적으로, 항체, 나노바디 및 DARPins와 같은 다른 스캐폴드는 면역화나 유도된 진화를 통해 다양한 표적에 대한 결합체로 개발됩니다 [36, 33, 12]. 하지만 이러한 실험적인 선택 방식은 표적 에피토프(특정 결합 위치)를 제어할 수 없으며, 일상적인 연구 응용에는 너무 번거로운 경우가 많습니다. 자연 단백질을 시작점으로 사용하지 않고 결합체를 de novo(새롭게) 설계하는 컴퓨터 설계 방법은 미리 지정한 에피토프를 목표로 하여 항체보다 더 작고, 열 안정성이 높으며, 발현이 쉬운 결합체를 생성할 수 있습니다 [10, 39, 51].
최근 딥러닝 기반 모델은 생체분자 구조 예측 [21, 2, 28, 24, 46]과 단백질 설계 [18, 73, 37, 14, 7, 34]에서 주요한 진전을 이루었습니다. 이는 단백질-단백질 상호작용의 예측과 설계 [9, 17, 4, 73, 11, 13, 53]와 같은 주요 과학적 및 사회적 과제의 진전을 가능하게 했습니다 [22]. 이제 고처리량 스크리닝 없이도 일부 표적에 대해 컴퓨터로 설계된 결합체를 얻을 수 있게 되었습니다 [73, 13, 11]. 작은 펩타이드나 비정형 표적과 같은 경우에는 실험적 최적화 없이도 높은 결합 친화도를 달성한 사례도 있습니다 [41, 44]. 하지만, 볼록하거나 극성 에피토프에 대한 성공률은 여전히 낮고, 초기 설계의 친화도는 보통 좋지 않으며, 많은 표적들이 여전히 다루기 어려운 상태로 남아 있습니다 [45, 3].
이 기술 보고서에서는 실험적 검증에 초점을 맞춰 AlphaProteo 단백질 설계 시스템을 소개하고, 이를 통해 다음과 같은 장점을 가진 de novo 단백질-결합 단백질을 설계할 수 있음을 보여줍니다:
- 높은 성공률: 안정적이고 발현이 잘 되며 특정성을 가지는 결합체를 수십 개의 설계 후보 중에서 선별하여 얻을 수 있어 고처리량 방법의 필요성을 줄일 수 있습니다.
- 높은 친화도: 테스트한 모든 표적에서 하나를 제외하고는 최상의 결합체가 서브 나노몰라 또는 낮은 나노몰라 수준의 결합 친화도(K_D)를 가지고 있어 추가적인 친화도 최적화 작업의 필요성을 최소화합니다.
- 범용성: 다양한 구조적 및 생화학적 특성을 가진 표적에 대해 단일 설계 방법만으로 복잡한 수동 개입 없이 결합체를 성공적으로 얻을 수 있습니다.
2. 결과
AlphaProteo는 두 가지 구성 요소로 이루어져 있습니다 (그림 1): Protein Data Bank (PDB)에서 구조 및 서열 데이터를 학습한 생성 모델과 AlphaFold 예측을 기반으로 한 증류 세트, 그리고 생성된 디자인이 실험적으로 성공할지를 예측하는 필터입니다. 결합체를 설계하기 위해 "표적" 단백질의 구조를 입력하고, 선택적으로 표적 에피토프를 나타내는 "핫스팟" 잔기를 지정합니다. 그러면 생성 모델은 해당 표적에 대한 후보 결합체의 구조와 서열을 출력합니다 (그림 1). 우리는 많은 수의 설계 후보를 생성하고, 실험적 테스트 이전에 더 작은 집합으로 필터링합니다. 이 생성 모델은 기존의 최고 방법들과 비교해 in silico 벤치마크에서 우수한 성과를 보였습니다 (그림 S1, 섹션 S2).
2.1 중간 처리량 스크리닝을 통한 서브 나노몰라 친화도의 결합체
AlphaProteo를 실험적으로 검증하기 위해, 우리는 다양한 구조적 특성을 가진 8개의 표적 단백질에 대해 결합체를 설계했습니다. 이 중 두 개는 감염과 관련된 바이러스 단백질이고, 나머지 여섯 개는 치료적으로 중요한 인간 단백질입니다 (그림 1, 표 S1):
- BHRF1: Epstein-Barr 바이러스에서 유래한 발암 단백질입니다. 이 단백질의 억제를 통해 암 세포를 죽이고 종양 성장을 늦출 수 있습니다 [66]. BHRF1은 결합 파트너의 헬릭스를 잘 수용할 수 있는 소수성 홈을 가지고 있어 결합을 촉진합니다.
- SARS-CoV-2 스파이크 단백질 수용체-결합 도메인 (SC2RBD): COVID-19 감염에 필요한 단백질 도메인입니다. 우리는 이 단백질이 인간 ACE2 수용체와 상호작용하는 인터페이스를 표적으로 삼았습니다. 이 상호작용을 방해하면 SARS-CoV-2가 인간 세포를 감염하는 것을 막을 수 있다는 것이 알려져 있습니다 [42]. 기존 설계 노력은 이 극성이고 볼록한 부위에 대해 성공했지만, 높은 친화도를 얻기 위해 실험적 최적화가 필요했습니다 [50, 11].
- 인터류킨-7 수용체-α (IL-7RA): 림프구 발달에 관여하고 급성 림프구성 백혈병 및 HIV의 치료적 표적인 세포 표면 수용체입니다. 우리는 IL-7의 결합 부위를 표적으로 삼았습니다. 이 부위는 중간 정도의 소수성을 가지고 있으며, 이전의 설계 노력에서 높은 성공률을 보였습니다 [51, 73].
- 프로그래밍된 사멸 리간드 1 (PD-L1): 면역 세포 증식을 조절하는 세포 표면 수용체로, 암 치료에 중요한 표적입니다. 이 표적 부위는 평평하며 작은 분자나 작은 단백질이 결합하기 어렵습니다 [11, 45].
- 트로포미오신 수용체 키나제 A (TrkA): 자가면역 질환에 관여하고 만성 통증 치료를 위한 진통제 표적인 신경 성장 인자 수용체입니다. 이전 설계 노력에서 다뤄진 소수성 포켓을 표적으로 삼았습니다. 이전의 결합 친화도는 실험적 최적화 없이 좋지 않았습니다 [51].
- 인터류킨-17A (IL-17A): 염증을 유발하는 분비 단백질로, 자가면역 질환에서 치료적 표적입니다. 우리는 IL-17A와 원래 수용체 간의 인터페이스를 표적으로 삼았습니다. 이는 동일한 단백질로 이루어진 두 개의 사슬을 포함하며 큰 극성 포켓을 가집니다. 기존의 설계된 IL-17A 결합체는 최적화되지 않은 친화도가 좋지 않았으며, 이를 얻기 위해 큰 라이브러리의 스크리닝이 필요했습니다 [3].
- 혈관 내피 성장 인자 A (VEGF-A): 혈관 생성을 조절하는 분비 성장 인자로, 암과 당뇨병성 망막병증의 치료적 표적입니다. 우리는 원래 VEGF 수용체가 결합하는 작은 소수성 부위를 표적으로 삼았습니다 [32]. 이 표적에 대한 설계된 결합체는 아직 출판된 바 없습니다.
- 종양 괴사 인자 알파 (TNFα): 염증 동안 생성되는 친염증성 사이토카인으로, 염증성 질환의 치료적 표적입니다 [16, 31, 30]. 우리는 TNFα 호모트리머의 두 서브유닛 사이의 극성 영역을 표적으로 삼았습니다. 이 표적에 대해 컴퓨터로 설계된 결합체는 보고된 바 없습니다.
우리는 이러한 표적들을 생물학적 중요성, 설계 문제의 난이도 범위를 포괄하기 위해, 그리고 기존 설계 방법들과 비교할 수 있도록 선택했습니다. RFdiffusion [73]과 비교하기 위해, 우리는 RFdiffusion이 가장 높은 실험적 성공률을 기록한 표적(IL-7RA)과 가장 낮은 성공률을 기록한 두 표적(PD-L1, TrkA)을 선택했으며, 실험 자원의 절약을 위해 나머지 두 개 표적은 제외했습니다. BHRF1과 SC2RBD는 각각 쉬운 표적과 어려운 표적으로 추가 선택했으며, 이는 기존 컴퓨터 설계 문헌에 기반합니다. IL-17A와 VEGF-A는 이 연구가 진행될 당시 컴퓨터 설계된 결합체가 확인되지 않았던 어려운 표적으로 선택되었습니다. 7개의 표적에 대한 실험적 테스트가 완료된 후, TNFα는 in silico 분석에 따라 8번째 매우 어려운 표적으로 선택되었습니다 (섹션 2.1.1, 섹션 S3). 이 연구 과정 동안 이 8개의 표적 이외의 추가적인 표적은 실험적으로 평가되지 않았습니다.

그림 1: AlphaProteo의 개요 및 실험적 성능
(A) 설계 시스템의 개략도. 생성 모델은 결합체 후보의 구조와 서열을 출력하고, 필터는 설계가 결합할지를 예측하는 모델 또는 절차입니다.
(B) 생성 모델이 수행한 표적 구조 기반 결합체 설계의 개략도입니다.
(C) 이번 연구의 결합체 설계 실험을 위해 사용된 7개 표적 단백질의 결정 구조(밝은 노란색)와 핫스팟 잔기(어두운 노란색 구체). VEGF-A와 IL-17A는 둘 다 이황화 결합된 동종이합체입니다. PDB ID 및 핫스팟 잔기 번호는 표 S1에서 참조하십시오.
(D) AlphaProteo(파란색) 또는 기존의 최고의 결합체 설계 방법(회색)에서 측정된 결합을 가진 모든 테스트된 설계의 비율입니다.
(E) AlphaProteo(파란색) 또는 기존의 최고 방법에서 타겟별 최상의 K_D 값. 이는 최적화되지 않은 컴퓨터 설계의 친화도를 나타냅니다 - 문헌에서 최적화된 컴퓨터 설계의 K_D 값은 표 1에서 참조하십시오.

표 1: AlphaProteo와 다른 방법들의 실험적 성공률과 친화도
AlphaProteo, RFdiffusion(효모 디스플레이를 사용한 측정, 섹션 S4 참조), 및 기타 컴퓨터 설계 방법에 대해 측정된 결합을 가진 설계의 비율과 표적별 최상의 결합체 친화도를 나열한 표입니다. 테스트된 설계의 수는 괄호 안에 표시되어 있습니다. "기타 설계 방법, 최적화"는 컴퓨터로 설계된 결합체의 실험적 최적화 후 얻어진 최상의 친화도를 나열합니다. 항체 및 나노바디와 같은 선택 기반 방법에서 파생된 결합체는 여기에서 고려하지 않았습니다. 문헌에서 가져온 K_D 값은 바이오레이어 간섭계(BLI) 또는 표면 플라즈몬 공명(SPR) 분석에서 나왔으며, 별표(*)로 표시된 경우 HTRF를 사용해 자체적으로 K_D를 측정했습니다 (섹션 S4 참조). 방법 개발에 사용된 일부 표적(섹션 S2)은 표 S2에서 더 자세한 결과를 가지고 있습니다.
-----
표는 AlphaProteo와 다른 결합체 설계 방법들이 8개의 표적 단백질에 대해 얼마나 성공적으로 결합체를 설계했는지, 그리고 그 결합체의 결합 친화도 (K_D 값)를 비교하고 있습니다. 두 가지 주요 지표는 실험적 성공률과 **결합 친화도 (K_D)**입니다.
1. 실험적 성공률 (%)
- 실험적 성공률은 설계된 결합체 중 실제로 실험에서 성공적으로 결합을 형성한 비율을 의미합니다. 이 값이 높을수록 더 많은 설계가 목표로 하는 단백질과 성공적으로 결합했다는 의미입니다.
- AlphaProteo, RFdiffusion, 그리고 기타 설계 방법들에 대해 각 표적에 대해 결과가 제공됩니다. 괄호 안의 숫자는 테스트된 설계의 수를 나타냅니다.
- BHRF1: 88% 성공률 (94개의 설계 중 88%가 성공)
- SC2RBD: 12% 성공률 (172개의 설계 중 12%가 성공)
- IL-7RA: 25% 성공률 (94개의 설계 중 25%가 성공)
- PD-L1: 15% 성공률 (159개의 설계 중 15%가 성공)
- TrkA: 9% 성공률 (131개의 설계 중 9%가 성공)
- IL-17A: 14% 성공률 (63개의 설계 중 14%가 성공)
- VEGF-A: 33% 성공률 (94개의 설계 중 33%가 성공)
- TNFα: 0% 성공률 (54개의 설계 중 아무 것도 성공하지 못함)
- IL-7RA: 17% 성공률 (95개의 설계 중 17%가 성공)
- PD-L1: 13% 성공률 (95개의 설계 중 13%가 성공)
- TrkA: 0.0% 성공률 (95개의 설계 중 아무 것도 성공하지 못함)
- BHRF1: 18% 성공률 (17개의 설계 중 18%가 성공)
- SC2RBD: 1.6% 성공률 (63개의 설계 중 1.6%가 성공)
- IL-7RA: 0.15% 성공률 (14,912개의 설계 중 0.15%가 성공)
- PD-L1: 13% 성공률 (16개의 설계 중 13%가 성공)
- TrkA: 0.07% 성공률 (14,982개의 설계 중 0.07%가 성공)
- IL-17A: 0.02% 성공률 (15,000개의 설계 중 0.02%가 성공)
- AlphaProteo:
AlphaProteo가 대부분의 표적에서 더 높은 성공률을 보이고 있습니다. 특히 BHRF1, IL-7RA, 그리고 VEGF-A에서 높은 성공률을 보이고 있으며, TNFα의 경우는 성공하지 못했습니다.
2. 결합 친화도 (K_D 값, nM)
- **결합 친화도 (K_D)**는 결합체가 얼마나 단단하게 표적 단백질에 결합하는지를 나타내는 값입니다. 이 값이 낮을수록 결합체가 표적에 더 강하게 결합합니다.
- BHRF1: 8.5 nM (94개의 설계 중)
- SC2RBD: 26 nM (172개의 설계 중)
- IL-7RA: 0.082 nM (94개의 설계 중) - 매우 높은 친화도
- PD-L1: 0.18 nM (159개의 설계 중)
- TrkA: 0.96 nM (131개의 설계 중)
- IL-17A: 8.4 nM (63개의 설계 중)
- VEGF-A: 0.48 nM (94개의 설계 중)
- TNFα: 해당 없음
- IL-7RA: 14 nM (95개의 설계 중) - 최적화되지 않은 상태에서 AlphaProteo보다 상대적으로 낮은 친화도
- PD-L1: 1.6 nM (95개의 설계 중)
- TrkA: 370 nM (95개의 설계 중) - 결합 친화도가 매우 낮음
- BHRF1: 58 nM (17개의 설계 중)
- SC2RBD: 100 nM (100,000개의 설계 중)
- IL-7RA: 3 nM (14,912개의 설계 중)
- PD-L1: 0.9 nM (16개의 설계 중)
- TrkA: 3000 nM (14,982개의 설계 중) - 친화도가 매우 낮음
- IL-17A: 47 nM (15,000개의 설계 중)
- AlphaProteo:
AlphaProteo는 대부분의 표적에 대해 낮은 K_D 값을 보여주며, 이는 높은 결합 친화도를 나타냅니다. 특히 IL-7RA와 VEGF-A에서 매우 낮은 K_D 값을 보이며, 이는 타겟 단백질과의 결합이 매우 강하다는 의미입니다. 반면, TrkA와 같은 표적에서 RFdiffusion 및 기타 설계 방법은 상대적으로 높은 K_D 값을 보였습니다.
요약:
- AlphaProteo는 높은 성공률과 우수한 결합 친화도를 보여주는 경향이 있습니다.
- 특히 IL-7RA, BHRF1, 그리고 VEGF-A와 같은 표적에 대해 AlphaProteo의 성능이 매우 뛰어납니다.
- RFdiffusion은 일부 표적에서 괜찮은 성과를 내지만, TrkA와 같은 특정 표적에서는 성공률이 낮고 친화도도 낮습니다.
- 기타 설계 방법들은 매우 낮은 성공률을 보이거나 최적화가 필요한 경우가 많습니다.
AlphaProteo의 성능은 기존의 최고 방법들과 비교해 상당히 우수하며, 이는 더 적은 실험적 최적화와 더 높은 성공 가능성을 의미합니다.
-----
2.1.1. 하나의 96-웰 플레이트 내에서 다수의 결합 히트 획득
각 표적에 대해 우리는 50-140개의 아미노산 길이의 많은 수의 in silico 설계를 생성하였고(Table S1), 자동 필터링 절차를 사용하여 효모 표면 디스플레이를 통해 결합을 테스트하기 위해 47개에서 172개의 결합체 후보를 선택했습니다. 우리는 7개의 초기 표적 세트를 대상으로 설계를 테스트했으며, 실험적 성공률(즉, 측정 가능한 결합을 보이는 설계의 비율, 섹션 S1.2)이 TrkA의 9%에서 BHRF1의 88%까지 다양하게 나타났습니다(Table 1). 표적별 성공률은 7개의 표적 중 5% 이상, 6개의 표적 중 10% 이상, 5개의 표적 중 20% 이상이었습니다(Figure 1, Table 1).
우리의 성공률은 7개의 표적에 대해 현재의 가장 우수한 대체 방법보다 높았습니다(Figure 2, Table 1). VEGF-A의 경우 AlphaProteo는 기존의 항체 개발 방법(예: 전통적인 방법으로 개발된 항체 [27]) 외에 최초로 성공적인 결합체를 얻은 컴퓨터 설계 방법입니다. BHRF1, SC2RBD, 그리고 IL-17A의 경우 AlphaProteo는 각각 다음으로 좋은 방법보다 5배, 8배, 700배 높은 성공률을 보였습니다(Figure 1, Table 1).
AlphaProteo를 현재의 최고 수준(State-of-the-Art, SoTA) 결합체 설계 방법인 RFdiffusion [73]과 정량적으로 비교하기 위해, 우리는 동일한 효모 디스플레이 실험에서 IL-7RA, PD-L1, TrkA에 대한 RFdiffusion의 결합체 설계와 AlphaProteo의 설계를 함께 테스트했습니다(섹션 S4). 이 직접 비교에서 AlphaProteo는 세 표적 모두에서 전반적으로 더 높은 실험적 성공률을 보였습니다(Figure 1, Table 1). 이러한 결과는 AlphaProteo가 성공률 측면에서 SoTA와 강력하게 경쟁할 수 있음을 나타냅니다.
우리는 SC2RBD, PD-L1, 그리고 TrkA가 AlphaProteo 개발에 사용되었기 때문에(섹션 S2), 이러한 성공률이 새로운 표적에 대한 성능을 과대평가할 수 있음을 언급합니다. 그러나 BHRF1, IL-7RA, VEGF-A, 그리고 IL-17A에 대해서는 단 한 번의 중간 처리량 테스트만 수행했으며, 이로 인해 비교적 어려운 표적에 대해서도 높은 성공률을 얻을 수 있음을 보여줍니다.
이 7개의 표적에 대한 결과를 얻은 후, 우리는 PDB에서 무작위로 선택된 200개의 표적 단백질에 대해 각 표적의 3개 에피토프에 대한 AlphaProteo의 in silico 성공률을 계산하여 AlphaProteo의 잠재적인 표적 범위를 조사했습니다(섹션 S3). 위 7개의 표적은 이 더 넓은 표적 리스트와 유사한 범위의 in silico 성공률을 보였으며, 이는 이들이 대부분의 잠재적 표적의 난이도를 대표함을 확인시켜 주었습니다. 이 스크리닝에서 TNFα를 포함하여 특히 어려운 표적들을 식별하였으며, 이들에 대한 in silico 성공률은 거의 0에 가까웠습니다. TNFα의 특이한 in silico 난이도와 높은 생의학적 중요성을 감안하여 이 표적에 대해 결합체를 설계하고 실험적으로 테스트했지만, 히트를 얻는 데 실패했습니다. 이는 이 표적에 대한 낮은 in silico 성능과 일치하며, 두 개의 서브유닛 사이의 평평하고 매우 극성인 결합 부위 때문일 가능성이 큽니다. 하지만 고무적인 점은 샘플링한 PDB 표적의 80%가 우리가 성공적으로 결합체를 얻은 가장 어려운 표적인 IL-17A보다 더 높은 in silico 성공률을 가지고 있다는 점입니다(Figure S2). 이는 AlphaProteo가 생물학적으로 중요한 다양한 결합체 설계 문제에 일반화될 수 있음을 시사합니다.
-----
AlphaProteo가 얼마나 넓은 범위의 표적 단백질에 대해 효과적으로 동작할 수 있는지를 확인하기 위해, PDB(Protein Data Bank)에서 무작위로 선택한 200개의 표적 단백질을 사용했습니다. 이 단백질들 각각에 대해 3개의 에피토프(단백질 상의 특정 결합 부위)를 설정하고, AlphaProteo가 컴퓨터 시뮬레이션(in silico)에서 얼마나 성공적으로 결합체를 설계할 수 있는지를 계산했습니다.
그 결과, AlphaProteo가 이 200개의 표적 단백질에 대해 보인 성공률 범위가 이전에 테스트했던 7개의 표적과 유사하다는 것을 알 수 있었습니다. 이는 AlphaProteo가 이 7개의 표적을 대표하는, 일반적인 난이도를 가진 표적들을 대상으로도 잘 동작할 수 있다는 것을 의미합니다.
이 스크리닝 과정에서 특히 어려운 표적도 몇 가지 확인되었는데, 그중 하나가 TNFα였습니다. 이 표적에 대해 AlphaProteo가 in silico에서 성공적으로 결합체를 설계할 가능성은 거의 0에 가까웠습니다. TNFα는 두 개의 서브유닛 사이의 평평하고 매우 극성인 결합 부위를 가지고 있어서 결합체 설계가 어려웠던 것으로 보입니다. 그럼에도 불구하고, 이 표적이 생의학적으로 중요한 단백질이라는 점에서 AlphaProteo를 사용해 실제 실험에서 결합체 설계를 시도했지만, 성공하지 못했습니다.
고무적인 점은, 우리가 샘플링한 PDB 표적의 80%가 AlphaProteo로 이미 성공한 가장 어려운 표적인 IL-17A보다 더 높은 성공률을 보였다는 것입니다. 즉, 대부분의 표적이 IL-17A보다 설계하기 쉽다는 뜻입니다. 이는 AlphaProteo가 생물학적으로 중요한 다양한 단백질에 대해 효과적으로 사용될 가능성이 크다는 것을 시사합니다.
간단히 요약하자면:
- AlphaProteo는 다양한 단백질에 대해 테스트했고, 대부분의 단백질에 대해 효과적인 결합체를 설계할 수 있는 가능성을 보였습니다.
- TNFα와 같이 결합 부위가 특이하고 어려운 표적도 있었지만, 대부분의 다른 표적은 IL-17A보다 성공률이 높았기 때문에 AlphaProteo가 여러 상황에 잘 적용될 가능성이 크다는 결론을 얻을 수 있습니다.
-----
2.1.2. 7개의 표적에 대한 최첨단 결합 친화도
높은 실험적 성공률은 결합체를 얻는 데 필요한 노동과 비용을 줄일 수 있지만, 히트를 발견한 후 더 중요한 지표는 표적에 대한 **결합 친화도 (K_D)**입니다. 대부분의 치료용 항체는 실험적 친화도 성숙 과정을 여러 번 거쳐서 낮은 피코몰라(pM) 수준의 K_D 값을 가집니다 [15, 40]. 연구 도구로 사용되는 결합체의 경우에도 낮은 나노몰라(nM) 수준 혹은 그 이상의 K_D 값이 일반적입니다 [26]. 우리가 설계한 결합체들이 표적에 얼마나 강하게 결합하는지를 측정하기 위해, 우리는 효모 스크리닝 히트를 재조합으로 E. coli에서 발현 및 정제하고, 그들의 K_D 값을 in vitro에서 측정했습니다. 전체적으로, 후속 연구를 위해 선택된 설계의 93%가 E. coli에서 성공적으로 발현되었으며 (Table S3), 대다수는 크기 배제 크로마토그래피에서 단분산(mono-disperse) 특성을 보였습니다 (Figure S4). 원형 이색성 (CD) 분광법으로 분석된 설계의 일부는 모두 예상된 2차 구조를 보였습니다 (Figure 2, Figure S5). 또한, 이 설계들은 CD 열 용융 테스트에서 최대 95°C까지 부분적 또는 전혀 펼쳐지지 않는 모습을 보여, 열 안정성(T_m 값) > 95°C 이상으로 매우 높음을 나타냈습니다 (Figure S5). 재조합 생산된 설계에 대해, 우리는 균질 시간 분해 형광 (HTRF) 평형 포화 결합 분석을 사용해 K_D 값을 측정했습니다 (섹션 S1.4).
AlphaProteo의 각 표적별 최상의 K_D 값은 4개의 표적에서 1 nM 미만, 6개의 표적에서 10 nM 미만, 그리고 7개의 표적에서 30 nM 미만이었습니다 (Figure 1, Table 1, Figure S3). 전체적으로 가장 좋은 K_D 값은 82 피코몰라(pM)로, 설계된 IL7RA_70에서 나왔습니다 (Table S7, Figure S6). 우리는 총 9개의 서브-나노몰라 K_D 값을 가진 결합체를 확인했습니다: IL-7RA에서 4개, PD-L1에서 2개, TrkA에서 1개, 그리고 VEGF-A에서 2개 (Table S7). 다른 설계 방법에서 얻은 최적화되지 않은 결합체와 비교했을 때, AlphaProteo의 K_D 값은 모든 표적에서 더 나았으며, BHRF1, SC2RBD, IL-7RA, PD-L1, TrkA, IL-17A에 대해 각각 7배, 4배, 37배, 5배, 380배, 5배 더 좋았습니다 (Figure 1, Table 1). 여러 번의 돌연변이와 선택 과정을 거쳐 실험적으로 최적화된 이전의 설계된 결합체와 비교했을 때도, AlphaProteo의 최상의 K_D 값은 BHRF1, IL-7RA, PD-L1, 그리고 TrkA에서 더 나았습니다 (Table 1, "기타 설계 방법, 최적화"). 이 모든 결과를 종합해 보면, AlphaProteo는 10-100개의 설계를 한 번만 스크리닝하고 추가적인 실험 없이도 많은 연구 응용에 사용할 수 있는 결합체를 생성할 수 있음을 시사합니다.
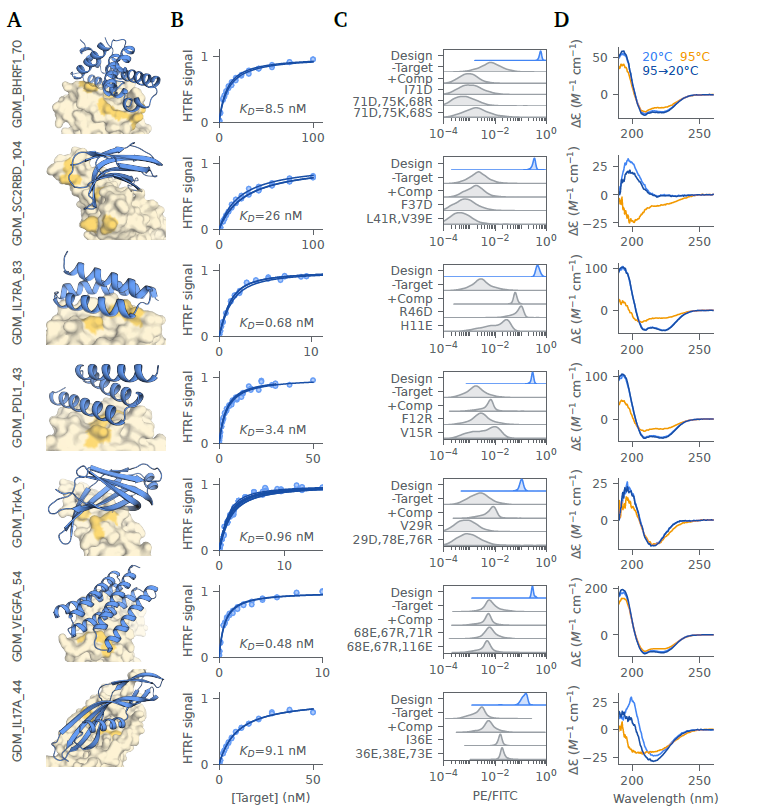
Figure 2: 각 표적에 대한 대표 결합체의 생화학적 특성 분석
(A) 설계 모델, (B) HTRF 평형 포화 결합 및 1:1 결합 모델에서 추정한 K_D 값, (C) 인터페이스 돌연변이에 대한 효모 디스플레이 및 경쟁 억제, 그리고 (D) 열 용융 전(20 ºC)과 후(95 ºC 및 95→20 ºC)의 원형 이색성 스펙트럼. 여기에서의 설계들은 네 가지 측정 유형을 모두 보여주기 위해 선택되었으며, 각 표적에 대한 가장 높은 친화도를 가진 결합체는 아닐 수도 있습니다. 각 표적에 대한 최상의 결합체와 그들의 K_D 값 목록은 Table S7에서 확인할 수 있습니다 (또한 Figure S6 참조). HTRF의 y축은 맞춰진 최대 신호에 정규화되어 있습니다 (추가 HTRF 데이터는 Figure S6에 있음).
2.1.3. 설계된 결합체가 의도한 에피토프에 결합함
설계된 결합체가 표적 단백질의 의도한 에피토프에 결합하는지 테스트하기 위해, 우리는 동일한 표적 부위에 결합하는 것으로 알려진 경쟁적 결합체가 있는 상태에서 결합을 측정했습니다 (그림 2, 섹션 S1). 예상대로 모든 경우에서 결합 신호가 감소했으며, 특히 우리의 결합체가 경쟁자보다 훨씬 높은 친화도를 가질 때 감소 폭이 작았습니다. 설계된 결합체가 의도한 상호작용을 통해 표적에 결합하는지 확인하기 위해, 우리는 설계 모델에서 표적 결합 인터페이스의 1-3개의 잔기를 돌연변이 시킨 후 최상위 결합체의 결합을 측정했습니다 (그림 2, 그림 S8, 그림 S9). 거의 모든 돌연변이는 부모 설계보다 낮은 결합 신호를 보였으며, 이는 돌연변이에 의해 결합 인터페이스가 성공적으로 방해되었음을 시사합니다. 소수의 돌연변이는 부모보다 더 높은 결합 신호를 보였는데, 이는 놀라운 일이 아닙니다. 우리는 돌연변이를 시각적 직관에 따라 선택했기 때문에, 구조적 미세 차이를 완전히 고려하지 못했을 가능성이 있어 결과적으로 결합이 개선될 수 있었던 것입니다 (섹션 S1.2.2). 전반적으로, 이 결과는 설계된 결합체와 표적이 의도된 인터페이스를 통해 서로 상호작용하고 있음을 나타냅니다.
2.1.4. 설계된 결합체는 특정한 표적에만 결합하며 구조적으로 다양함
우리가 설계한 최상위 결합체의 일부에 대한 특이성을 테스트하기 위해, 이들이 7개의 표적에 대해 각각 어떻게 결합하는지 측정했습니다. 테스트된 모든 결합체는 의도된 표적에 대해서만 결합 신호를 보였으며 (그림 3), 이는 많은 후속 응용에서 더욱 철저한 특이성 테스트(예: 모든 프로테오믹 표적에 대한 테스트)가 필요할 수 있다는 점을 고려해야 합니다.
우리는 성공적인 설계의 구조적 다양성을 분석하여, 각 설계 문제에 대해 우리 방법이 얼마나 많은 독립적인 해결책을 생성할 수 있는지에 대한 통찰을 얻고자 했습니다. 구조적 다양성은 또한 실질적으로 중요한데, 이는 사전에 알 수 없는 후속 요구사항을 만족시킬 수 있는 설계가 나올 가능성을 최대화하기 때문입니다. 우리는 각 표적에 대한 결합 히트에서 쌍(pair)별 TM-score의 분포 (그림 S10)와 2차 구조 내용(그림 S10)을 살펴보았습니다. RFdiffusion에서 생성된 활성 설계들과 비교했을 때, AlphaProteo의 설계들은 서로 구조적 유사성이 더 낮고, all-beta 구조의 빈도가 더 높았습니다. 이러한 관찰은 실험적으로 확인된 결합체 설계를 시각적으로 검사한 결과와도 일치하며, 이는 all-alpha, 혼합 alpha/beta, 그리고 all-beta 접힘 구조의 다양한 모습을 보여줍니다 (그림 3).
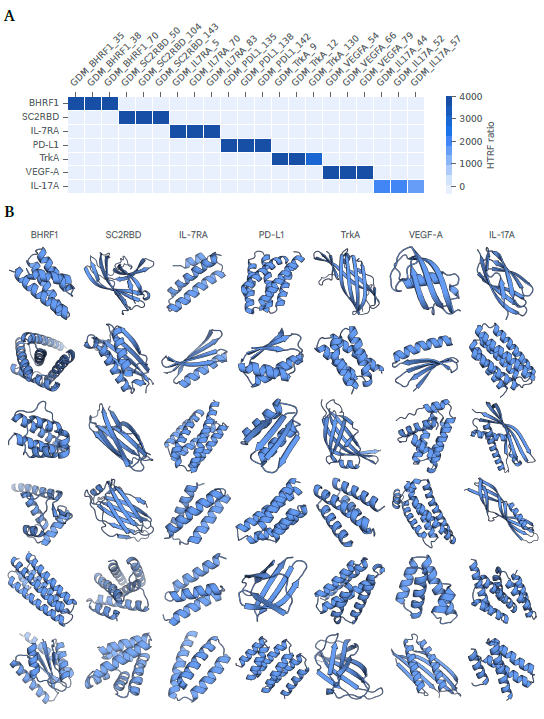
그림 3: 설계된 결합체의 특이성과 다양성
(A) 특이성: 상위 결합체(1 nM)의 일부에 대해 각 표적(100 nM)에 대해 측정한 HTRF 결합 신호. 모든 결합체는 표적에 대한 결합 신호를 보여주며, 테스트된 오프-타겟들에 대해서는 비특이적 결합 신호가 없습니다.
(B) 다양성: TM-score 컷오프 0.6에서 서로 다른 구조적 클러스터에서 실험적으로 확인된 AlphaProteo 결합체의 예시.
2.2 결합체의 기능적 및 구조적 검증
2.2.1 결합체가 라이브 바이러스 중화 실험에서 SARS-CoV-2 변이체를 중화함
우리의 결합체가 의도된 생물학적 활동을 나타내는지 확인하기 위해, 우리는 이들이 살아 있는 SARS-CoV-2에 결합하고 이를 중화하는 능력을 테스트했습니다. 우리는 네 개의 결합체(GDM_SC2BRD_11, GDM_SC2BRD_27, GDM_SC2RBD_104, GDM_SC2BRD_50)를 사용해 2020년부터 2024년까지 전 세계적으로 유행한 SARS-CoV-2의 네 가지 변이체를 Vero 세포에 감염시키지 못하도록 중화할 수 있는지 시험했습니다 [71]. 네 개의 결합체 모두 조상 변이(hCoV19/England/02/2020)를 성공적으로 중화했으며, 50% 억제 농도(EC50)는 89-300 nM 범위였습니다 (그림 4, 그림 S11). 이 변이체는 2019년에 처음 확인된 바이러스와 동일한 스파이크 단백질을 가지고 있으며, 설계에 사용된 표적 구조의 출처이기도 합니다. 이러한 EC50 값은 실험실에서 측정된 결합 친화도(in vitro)보다 2배에서 10배 높은데, 이는 임상용 단클론 항체(예: sotrovimab)의 동일한 시험에서 관찰된 결과와 일치합니다 (K_D=0.21 nM, EC50=0.67 nM, 단일 SARS-CoV-2 변이에 대해) [71].
흥미롭게도, 네 개의 결합체 중 두 개(GDM_SC2RBD_11 및 GDM_SC2RBD_129)는 테스트된 세 개의 변이체를 중화할 수 있었습니다. 가장 강력한 효과를 보였고 가장 낮은 EC50 값을 가진 결합체(GDM_SC2RBD_50)는 조상 변이체만을 억제했습니다. 네 가지 변이체 모두 최소한 하나 이상의 설계된 결합체에 의해 중화되었습니다.
2.2.2 결합체가 VEGF 수용체의 하위 신호 전달을 억제함
우리는 또한 설계된 결합체 GDM_VEGFA_54와 GDM_VEGFA_71이 VEGF 신호를 억제하는 능력을 테스트했습니다. 우리는 인간 VEGF-A로 자극된 1차 인간 제대정맥 내피세포(HUVECs)에서 **VEGF 수용체 2 (VEGFR2)**와 하위 ERK 및 AKT 키나제의 인산화를 측정했습니다 (그림 4). GDM_VEGFA_54와 함께 배양한 결과, ERK, AKT, 및 VEGFR2의 인산화가 VEGF-A만 있는 대조군("억제제 없음")과 비교해 상당히 감소했습니다 (그림 4). 이러한 효과는 강력한 소분자 VEGFR2 키나제 억제제인 ki8751 [25]의 효과와 유사합니다. 이 효과는 임상에서 승인된 약물 Avastin의 활성 성분인 항-VEGF-A 단클론 항체 베바시주맙의 효과보다 더 강력했으며, 이 실험에서 베바시주맙을 결합체와 동일한 몰 농도로 테스트했습니다. 이 농도는 HUVECs에서 in vitro로 보통 테스트되는 농도보다 1000배 낮은 농도이므로, GDM_VEGFA_54가 HUVECs에서 베바시주맙보다 더 강력한 VEGF-A 억제제임을 시사합니다. 두 번째로 테스트된 결합체 GDM_VEGFA_71은 ERK, AKT, 및 VEGFR2의 인산화가 감소하는 효과를 보였지만, 그 감소 효과는 GDM_VEGFA_54보다는 약했습니다. 이러한 결과는 VEGF-A에 대한 GDM_VEGFA_54와 GDM_VEGFA_71의 in vitro 결합 친화도가 각각 0.48 nM 및 4.7 nM임과 일치합니다.
2.2.3 결합체-표적 복합체의 실험적 구조가 결합 모드와 구조를 확인함
우리의 설계된 결합체의 구조와 결합 모드를 검증하기 위해, 우리는 극저온 전자현미경(cryo-EM)을 사용해 GDM_SC2RBD_11, GDM_SC2RBD_50, GDM_SC2RBD_104, 그리고 GDM_SC2RBD_129가 SARS-CoV-2 스파이크 S1 단백질과 결합한 복합체의 구조를 4.5 - 6.0 Å 해상도로 얻었습니다 (그림 5 및 그림 S12). 실험적으로 얻은 구조는 설계된 결합체-표적 복합체의 구조와 매우 유사했으며, 스파이크 S1 단백질을 기준으로 정렬했을 때 결합체의 Cα RMSD는 0.84 - 3.14 Å 범위였습니다. 추가로, 우리는 VEGF-A와 결합한 GDM_VEGFA_71의 X선 결정 구조를 2.65 Å 해상도로 얻었습니다 (그림 5). 결합체는 설계된 구조와 매우 밀접하게 접힘을 형성했으며, 이는 혼합된 알파-베타 구조로 5가닥의 베타 시트가 VEGF-A와 상호작용하고 있었습니다. 실험적으로 확인된 구조와 AF3 모델 간의 Cα RMSD는 0.78 Å로 원자 수준의 정확도를 보여주었습니다. 또한, 설계된 결합 방향도 매우 정확했으며, 표적에 정렬된 결합체의 Cα RMSD는 1.65 Å였습니다. 표적과 상호작용하는 결합체의 대부분의 사이드체인은 정확한 로타머를 가지고 있었으며, 여기에는 결합체의 히스티딘과 VEGF-A의 티로신 사이의 묻힌 수소 결합이 포함되어 있으며, 이는 실험 구조에서 거의 완벽하게 재현되었습니다 (그림 5).
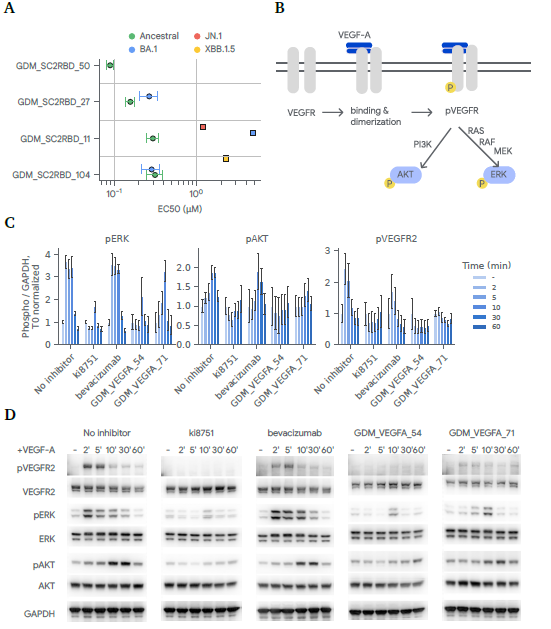
그림 4: 설계된 결합체에 의한 SARS-CoV-2 바이러스 감염 및 VEGF 신호 억제
(A) SARS-CoV-2의 4가지 변이에 대해 바이러스 중화 시험에서 4가지 설계된 SC2RBD 결합체의 50% 억제 농도(EC50). 에러 막대는 기본 용량-반응 곡선의 표준 오차를 보여줍니다. 완전한 중화(0% 감염)가 관찰되지 않은 저친화도 결합체는 사각형 기호로 표시됩니다. 이러한 경우 용량-반응 곡선에 대한 EC50 추정치의 에러를 의미 있게 결정할 수 없어 에러 막대는 생략되었습니다.
(B) VEGF-A 신호 전달 경로의 개략도. VEGF-A의 결합은 VEGFR의 이량체화, VEGFR의 인산화 및 하위 신호 전달 경로를 유발하여 ERK와 AKT를 인산화시킵니다.
(C) 소분자 VEGFR2 억제제 ki8751, 단클론 항체 베바시주맙, 또는 설계된 VEGF-A 결합체로 처리한 후의 인산화된 ERK, AKT, 및 VEGFR2 웨스턴 블롯 밴드 강도의 비율(전처리 전(-) 및 처리 후 2, 5, 10, 30, 60분). 값은 전처리 값으로 정규화되었습니다. 보여지는 값은 3회(결합체) 또는 6회(대조군)의 생물학적 반복 실험의 평균 및 표준 오차입니다.
(D) VEGF-A와 결합체 GDM_VEGFA_54, GDM_VEGFA_71, ki8751, 또는 베바시주맙으로 2분에서 60분간 처리한 후 HUVEC 세포에서의 인산화된 ERK, AKT, 및 VEGFR2의 웨스턴 블롯. VEGF-A 신호 억제는 pERK, pAKT, 및 pVEGFR2 밴드 강도의 감소로 관찰됩니다. 이는 VEGF-A만 사용한 대조군("억제제 없음")과 비교한 결과입니다.
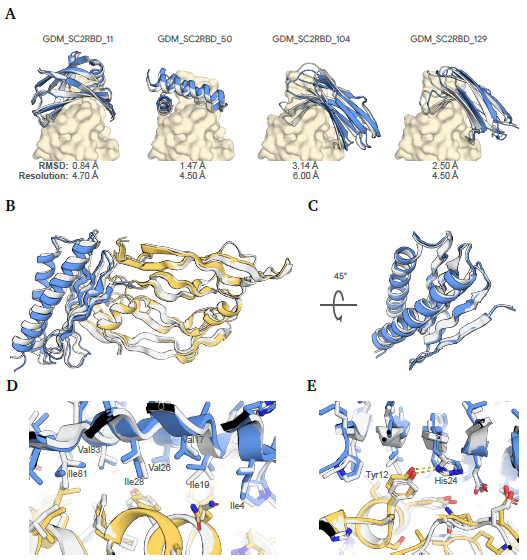
그림 5: SARS-CoV-2 스파이크 및 VEGF-A에 대한 결합체의 실험적 구조
(A) SARS-CoV-2 스파이크 단백질(노란색)과 결합한 설계된 결합체(파란색)의 극저온 전자현미경(cryo-EM) 구조로, 스파이크 단백질에 AF2-multimer 예측(회색)과 정렬되었습니다. Cryo-EM 구조 해상도와 AF2-multimer와 실험적 구조 간의 표적에 정렬된 결합체 Cα RMSD 값이 표시되어 있습니다.
(B) VEGF-A 동종이합체(노란색)와 설계된 GDM_VEGFA_71(파란색)의 복합체 결정 구조로, VEGF-A에 AF2-multimer 예측(회색)과 정렬되었습니다 (결합체 Cα RMSD = 1.65 Å).
(C) 결합체 단량체의 회전된 뷰(결합체 정렬된 결합체 Cα RMSD = 0.78 Å).
(D-E) 실험적 구조와 AF2-multimer 예측 설계 사이의 사이드체인의 근접 일치를 보여주는 결합체-표적 인터페이스의 근접 보기.
(D) 인터페이스에서 결합체의 소수성 사이드체인 포장. 대부분의 경우 설계와 구조 사이의 일치가 거의 완벽하지만, Val17, Ile19, Ile81의 경우 약간의 차이가 있습니다.
(E) 설계된 수소 결합은 결합체의 His24와 VEGF-A의 Tyr12 사이에 존재합니다.
3. 결론
우리의 결과는 AlphaProteo가 단일 라운드의 중간 처리량 테스트 후 다양한 표적에 대해 낮은 나노몰라부터 서브 나노몰라 수준의 결합체를 생성할 수 있음을 보여줍니다. 이 결합체들은 크기가 작고(5-15 kDa), 열 안정성이 있으며, 고발현되기 때문에 추가적인 최적화 없이도 일부 연구 응용에서 바로 사용될 가능성이 있습니다. 하지만, 이번 연구에서는 비교적 적은 수의 표적만 실험적으로 검증하였으며, 모든 결합체가 표적의 결정 구조를 입력으로 사용해 설계되었다는 점을 주목해야 합니다. 우리는 AlphaProteo의 성능을 더욱 향상시키고 TNFα와 같은 어려운 표적, 그리고 실험적 구조가 없거나 하나의 잘 정의된 형태가 없는 표적들을 포함하여 더 넓은 범위의 결합체 설계 문제를 해결할 수 있는 능력을 확장하고자 합니다. 우리는 AlphaProteo가 세포 신호 조절, 단백질, 세포, 조직의 이미징, 다양한 효소 시스템에 표적 특이성을 부여하는 것 등 많은 생물학적 응용에 새로운 해결책을 제공할 것으로 믿고 있습니다.
추가 주의 사항
이 보고서의 내용은 연구 목적으로만 사용되며, 임상용으로 사용될 수 없습니다. 본 보고서에는 생명 안전 및 상업적 고려로 인해 기계 학습 방법이 포함되어 있지 않습니다. 우리는 생명 안전과 안전성에 대한 연구와 자문을 바탕으로, 커뮤니티를 위한 안전하고 책임 있는 단백질 설계 솔루션을 개발하려고 노력하고 있습니다.
감사의 말
저자들은 다음 사람들의 조언과 피드백에 감사드립니다: Jonas Adler, Andy Ballard, Charlie Beattie, David Belanger, Lucy Colwell, Andrew Cowie, Sarah Elwes, Richard Evans, Conor Griffin, John Jumper, Svend Kjær, Antonia Paterson, Matteo Perino, Francesca Pietra, Uchechi Okereke, Olaf Ronneberger, Freyr Sverrisson, Nick Swanson, Kathryn Tunyasuvunakool, Augustin Žídek. 또한, Dane Wittrup (매사추세츠 공과대학 화학공학과)에게 효모 벡터 pCTcon2를 후하게 제공해 주신 것에, 그리고 Svend Kjær (프랜시스 크릭 연구소 구조 생물학 과학 기술 플랫폼)에게 SARS-CoV-2 스파이크 단백질의 생산에 대해 감사를 표합니다.
기여 사항
기계 학습 모델 개발, 설계 후보 생성, 실험적 성공률, 실험적 결합 친화도 측정, 및 VEGF-A 결합체의 결정 구조 결정은 Google DeepMind에 의해 수행되었습니다.
세포 기반 분석 및 극저온 전자현미경(cryo-EM) 구조 결정은 영국 런던의 프랜시스 크릭 연구소의 연구 그룹에 의해 수행되었습니다.
'인공지능' 카테고리의 다른 글
| Pixtral 12B (1) | 2024.11.23 |
|---|---|
| DeepSeek-V2.5 (1) | 2024.11.23 |
| AlphaProteo generates novel proteins for biology and health research (1) | 2024.11.22 |
| VASA-1: Lifelike Audio-Driven Talking FacesGenerated in Real Time (3) | 2024.11.21 |
| EMO: Emote Portrait Alive -- Generating Expressive Portrait Videos with Audio2Video Diffusion Model under Weak Conditions (4) | 2024.11.21 |


