https://arxiv.org/abs/2311.18803
BioCLIP: A Vision Foundation Model for the Tree of Life
Images of the natural world, collected by a variety of cameras, from drones to individual phones, are increasingly abundant sources of biological information. There is an explosion of computational methods and tools, particularly computer vision, for extra
arxiv.org
초록 (Abstract)
드론부터 개인 휴대폰까지 다양한 카메라로 수집된 자연 세계의 이미지는 점점 더 풍부한 생물학적 정보의 원천이 되고 있습니다. 특히 컴퓨터 비전 분야에서는 과학 및 보존 목적으로 이미지에서 생물학적으로 관련된 정보를 추출하기 위한 계산 방법과 도구가 폭발적으로 증가하고 있습니다. 하지만 대부분의 방법은 특정 작업을 위해 설계된 맞춤형 접근 방식이며 새로운 질문, 맥락 및 데이터 세트에 쉽게 적용하거나 확장할 수 없습니다. 이미지에 대한 일반적인 생물 질문에 대한 비전 모델이 시급히 필요합니다. 이를 위해, 우리는 가장 크고 다양한 ML-ready 생물 이미지 데이터 세트인 TREEOFLIFE-10M을 선별하고 공개합니다. 그런 다음 TREEOFLIFE-10M에 의해 포착된 생물학의 고유한 특성, 즉 풍부하고 다양한 식물, 동물 및 균류 이미지와 풍부한 구조화된 생물학적 지식의 가용성을 활용하여 생명의 나무를 위한 기초 모델인 BIOCLIP을 개발합니다. 다양한 세분화된 생물학 분류 작업에 대한 엄격한 벤치마킹을 통해 BIOCLIP이 기존 기준선보다 지속적으로 훨씬 뛰어난 성능을 발휘한다는 사실을 발견했습니다 (절대적으로 16%~17%). 본질적인 평가 결과 BIOCLIP은 생명의 나무에 부합하는 계층적 표현을 학습하여 강력한 일반화 능력에 대한 통찰력을 제공한다는 것을 보여줍니다.
1. 서론
디지털 이미지와 컴퓨터 비전은 진화 생물학[13, 51]부터 생태학 및 생물 다양성[5, 77, 83]에 이르기까지 자연 세계를 연구하는 데 널리 사용되는 도구가 되고 있습니다. 박물관[64], 카메라 트랩[1, 6, 7, 59, 77], 시민 과학 플랫폼[2, 40, 54, 58, 60, 62, 75,79–81, 87, 88]에서 얻은 방대한 양의 이미지를 종 분류, 개체 식별 및 특성 감지와 같은 실행 가능한 정보로 신속하게 변환하는 기능은 종 식별[32], 적응 메커니즘 이해[23, 39], 풍부함 및 개체군 구조 추정[3, 40, 58, 82], 생물 다양성 모니터링 및 보존[83]과 같은 작업에서 새로운 발전을 가속화하고 가능하게 했습니다.
그러나 컴퓨터 비전을 적용하여 생물학적 질문에 답하는 것은 여전히 상당한 기계 학습 전문 지식과 노력을 필요로 하는 힘든 작업입니다. 생물학자들은 관심 있는 특정 분류군과 작업에 대해 충분한 데이터에 수동으로 라벨을 지정하고 작업에 적합한 모델을 찾아 훈련해야 합니다. 한편, CLIP[69] 및 GPT-3[14]와 같은 기초 모델[12]은 광범위한 작업에 대해 zero-shot 또는 few-shot 학습을 가능하게 함으로써 매우 가치가 있습니다. 생물학을 위한 유사한 비전 기초 모델은 훈련된 분류군뿐만 아니라 전체 생명의 나무[37, 53]에 걸쳐 작업에 유용해야 합니다. 이러한 모델은 AI를 생물학에 적용하는 데 드는 장벽을 크게 낮출 것입니다.
본 연구에서는 생명의 나무를 위한 이러한 비전 기초 모델을 개발하는 것을 목표로 합니다. 실제 생물학 작업에 널리 유용하려면 이 모델은 다음 기준을 충족해야 합니다. 먼저, 가능한 경우 생명의 나무 전체로 일반화하여 틈새가 아닌 다양한 클래스를 연구하는 연구자를 지원해야 합니다. 또한, 수백만 개의 알려진 분류군[38, 44]을 포괄하는 훈련 데이터를 수집하는 것은 불가능하므로 모델은 훈련 데이터에 없는 분류군으로 일반화해야 합니다. 둘째, 생물학은 동일한 속 내에서 밀접하게 관련된 종[67]이나 적합성 이점을 위해 다른 종의 모습을 모방하는 종[39]과 같이 시각적으로 유사한 유기체를 자주 다루기 때문에 유기체 이미지에 대한 세분화된 표현을 학습해야 합니다. 이러한 세분화된 세분성은 생명의 나무가 생물을 광범위한 범주(동물, 균류 및 식물)와 매우 세분화된 범주(그림 1 참조) 모두로 구성하기 때문에 매우 중요합니다. 마지막으로, 생물학에서 데이터 수집 및 레이블 지정 비용이 많이 들기 때문에 저데이터 영역(즉, zero-shot 또는 few-shot)에서 강력한 성능이 중요합니다.
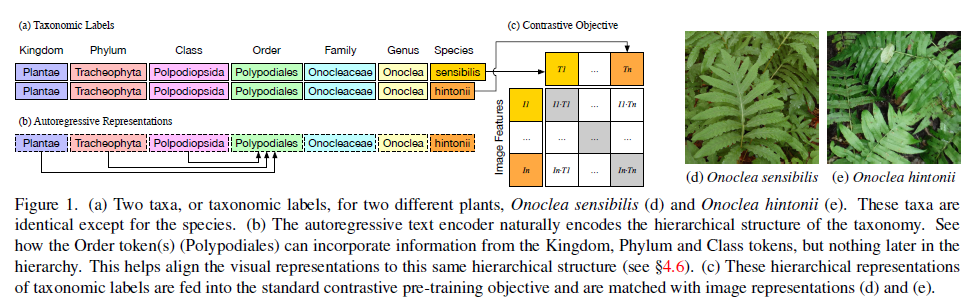
그림 1. (a) 두 가지 식물, Onoclea sensibilis (d) 및 Onoclea hintonii (e)에 대한 두 가지 분류군 또는 분류 레이블. 이러한 분류군은 종을 제외하고는 동일합니다. (b) 자기 회귀 텍스트 인코더는 자연스럽게 분류 체계의 계층 구조를 인코딩합니다. Order 토큰(Polypodiales)이 Kingdom, Phylum 및 Class 토큰의 정보를 통합할 수 있지만 계층 구조의 후반부에는 아무것도 통합할 수 없는 방법을 확인하십시오. 이렇게 하면 시각적 표현을 동일한 계층적 구조에 맞출 수 있습니다(§4.6 참조). (c) 이러한 분류 레이블의 계층적 표현은 표준 대조 사전 훈련 목표에 입력되고 이미지 표현과 일치합니다(d 및 e).
일반화, 세분화된 분류, 데이터 효율성이라는 목표는 컴퓨터 비전에서 새로운 것은 아니지만, 수억 개의 이미지로 훈련된 기존의 일반 영역 비전 모델[61, 69, 95]은 진화 생물학 및 생태학에 적용될 때 부족함을 드러냅니다. 특히, 기존의 비전 모델은 개와 늑대처럼 흔한 유기체를 비교하는 데 유용한 일반적인 세분화된 표현을 생성하지만, Onoclea sensibilis와 Onoclea hintonii와 같은 더 세분화된 비교에는 적합하지 않습니다(그림 1 참조).
저희는 생물학을 위한 비전 기초 모델 개발에 두 가지 주요 장벽을 확인했습니다. 첫째, 적합한 사전 훈련 데이터 세트가 필요합니다. 기존 데이터 세트[28, 86, 88, 89]에는 규모, 다양성 또는 세분화된 레이블이 부족합니다. 둘째, 생명의 나무 분류 체계와 같이 주류 사전 훈련 알고리즘[48, 61, 69]에서 충분히 고려되지 않은 생물학 영역의 특수 속성을 활용하여 세 가지 중요한 목표를 더 잘 달성할 수 있는 적절한 사전 훈련 전략을 조사해야 합니다.
이러한 목표와 이를 달성하는 데 있어 어려움을 고려하여, 우리는 1) 대규모 ML-ready 생물 이미지 데이터 세트인 TREEOFLIFE-10M과 2) TREEOFLIFE-10M에서 분류군을 적절히 사용하여 훈련된 생명의 나무를 위한 비전 기초 모델인 BIOCLIP을 소개합니다. 아래에서 기여도, 개념적 프레임워크 및 설계 결정을 간략하게 설명합니다.
TREEOFLIFE-10M: 대규모의 다양한 ML-ready 생물 이미지 데이터 세트입니다. 관련 분류 레이블이 있는 생물 이미지의 현재까지 가장 큰 ML-ready 데이터 세트를 선별하고 공개하며, 생명의 나무에서 454,000개의 분류군을 포괄하는 1,000만 개 이상의 이미지를 포함합니다.2 현재 가장 큰 ML-ready 생물 이미지 데이터 세트인 iNat21 [86]은 10,000개의 분류군을 포괄하는 270만 개의 이미지만 포함합니다. TREEOFLIFE-10M은 iNat21 및 BIOSCAN-1M [28]과 같은 기존 고품질 데이터 세트를 통합합니다. 더 중요한 것은 TREEOFLIFE-10M의 데이터 다양성의 대부분을 제공하는 Encyclopedia of Life (eol.org)의 새로 선별된 이미지를 포함한다는 것입니다. TREEOFLIFE-10M의 모든 이미지에는 가능한 가장 세밀한 수준의 분류 계층 구조와 생명의 나무에서 더 높은 분류 순위가 레이블로 지정되어 있습니다(분류 순위 및 레이블의 예는 그림 1 및 표 3 참조). TREEOFLIFE-10M을 사용하면 BIOCLIP 및 향후 생물학 기초 모델을 교육할 수 있습니다.
BIOCLIP: 생명의 나무를 위한 비전 기초 모델입니다. TREEOFLIFE-10M과 같은 대규모 레이블이 지정된 데이터 세트를 사용하면 표준적이고 직관적인 교육 전략(ResNet50 [33] 및 Swin Transformer [48]과 같은 다른 비전 모델에서 채택)은 감독 분류 목표를 사용하고 이미지에서 분류 지수를 예측하는 방법을 배우는 것입니다. 그러나 이것은 풍부한 분류 레이블 구조를 인식하고 활용하지 못합니다. 분류군은 고립되어 존재하는 것이 아니라 포괄적인 분류 체계에서 상호 연결되어 있습니다. 결과적으로 일반 감독 분류를 통해 훈련된 모델은 훈련에서 보이지 않는 분류군에 잘 일반화되지 않을 수 있으며 보이지 않는 분류군의 zero-shot 분류를 지원하지 못할 수도 있습니다.
대신, 우리는 CLIP 스타일의 다중 모드 대조 학습[69]과 BIOCLIP을 위한 풍부한 생물학적 분류를 결합한 새로운 전략을 제안합니다. Kingdom에서 가장 먼 분류군 순위까지 분류 체계를 분류 이름이라고 하는 문자열로 "평평하게" 만들고 CLIP 대조 학습 목표를 사용하여 이미지를 해당 분류 이름과 일치시키는 방법을 학습합니다. 직관적으로 이것은 모델이 보이지 않는 분류군으로 일반화하는 데 도움이 됩니다. 모델이 종을 본 적이 없더라도 해당 종의 속이나 과에 대한 합리적인 표현을 학습했을 가능성이 높습니다(그림 1 참조). BIOCLIP은 보이지 않는 분류군의 분류 이름으로 zero-shot 분류도 지원합니다. 우리는 또한 혼합 텍스트 유형 훈련 전략을 제안하고 그 효과를 입증합니다. 훈련 중에 서로 다른 텍스트 유형(예: 분류학적 이름 vs. 과학적 이름 vs. 일반 이름)을 혼합함으로써 분류학적 이름에서 얻은 일반화를 유지하면서 테스트 시 더 많은 유연성을 얻습니다. 예를 들어 BIOCLIP은 다운스트림 사용자가 일반적인 종 이름만 제공하더라도 여전히 뛰어납니다.
포괄적인 벤치마킹: 훈련에서 보이지 않는 새로 선별된 RARE SPECIES 데이터 세트를 포함하여 동물, 식물 및 균류를 포괄하는 10개의 세분화된 이미지 분류 데이터 세트에서 BIOCLIP을 포괄적으로 평가합니다. BIOCLIP은 zero-shot 및 few-shot 설정 모두에서 강력한 성능을 달성하고 CLIP [69] 및 OpenCLIP [42]보다 훨씬 뛰어난 성능을 발휘하여 평균 절대 개선율 17%(zero-shot) 및 16%(few-shot)를 달성합니다. 내재적 분석은 BIOCLIP이 생명의 나무에 부합하는 더 세분화된 계층적 표현을 학습하여 우수한 일반화를 설명한다는 것을 추가로 보여줍니다.
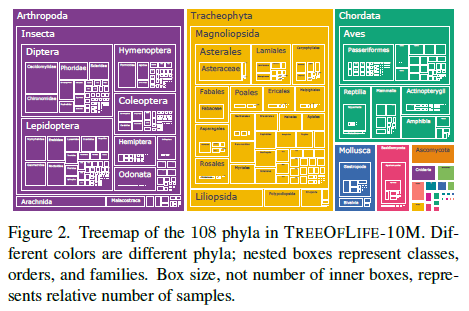
그림 2. TREEOFLIFE-10M의 108개 문의 트리맵. 서로 다른 색상은 서로 다른 문을 나타냅니다. 중첩된 상자는 클래스, 주문 및 가족을 나타냅니다. 내부 상자의 수가 아니라 상자 크기는 상대적인 샘플 수를 나타냅니다.
최근 연구에 따르면 CLIP 모델 훈련 시 데이터 품질과 다양성이 중요한 것으로 나타났습니다 [24, 26, 57]. 우리는 생물학 분야의 컴퓨터 비전 모델을 위한 가장 다양한 대규모 공개 ML-ready 데이터 세트인 TREEOFLIFE-10M을 선별했습니다.
2.1. 이미지
가장 큰 ML-ready 생물 이미지 데이터 세트는 10K 종의 270만 개 이미지를 포함하는 iNat21 [86]입니다. ImageNet-1K [70]와 같은 인기 있는 일반 도메인 데이터 세트와 비교하여 이 클래스 폭은 넓지만, 생물학에서는 10K 종은 제한적입니다. 국제 자연 보전 연맹(IUCN)은 2022년에 200만 종 이상의 총 설명된 종을 보고했으며, 조류 종만 10K 종 이상, 파충류 종만 10K 종 이상입니다[44]. iNat21의 종 다양성은 생명의 나무 전체를 위한 기초 모델 훈련 가능성을 제한합니다.
종 다양성에 중점을 둔 고품질 생물 이미지를 찾기 위해 Encyclopedia of Life 프로젝트(EOL; eol.org)를 살펴봅니다. EOL은 다양한 기관과 협력하여 수백만 개의 이미지를 수집하고 레이블을 지정합니다. EOL에서 660만 개의 이미지를 다운로드하고 데이터 세트를 확장하여 추가로 440K 분류군을 다룹니다.
종은 생명 나무의 다른 하위 트리에 균등하게 분포되어 있지 않습니다. 곤충(100만 종 이상의 Insecta 클래스), 조류(10K 종 이상의 Aves 클래스) 및 파충류(10K 종 이상의 Reptilia 클래스)는 훨씬 더 많은 종을 가진 매우 다양한 하위 트리의 예입니다. 기초 모델이 곤충에 대한 매우 세분화된 시각적 표현을 학습할 수 있도록 494개의 서로 다른 계통을 포괄하는 100만 개의 곤충 실험실 이미지에 대한 최근 데이터 세트인 BIOSCAN-1M [28]도 통합합니다.3 또한 BIOSCAN-1M에는 iNat21과 같은 현장 이미지가 아닌 실험실 이미지가 포함되어 있어 이미지 분포가 다양해집니다.
2.2. 메타데이터 및 집계
TREEOFLIFE-10M 데이터 세트는 iNat21(훈련 분할), 선별된 EOL 데이터 세트 및 BIOSCAN-1M을 이미지를 집계하고 레이블을 정규화하여 통합합니다. 이것은 매우 중요하지 않은 작업입니다. 분류 계층 구조는 악명 높게 노이즈가 많고 출처 간에 거의 일치하지 않기 때문에 [4, 31, 36, 52, 63] 생명의 나무 전체에 대한 기초 규모 비전 모델을 훈련하기에 충분히 큰 이미지 데이터 세트가 이전에 부족했던 원인이 되었을 가능성이 큽니다. 우리는 동음이의어(상위 분류군 간에 공유되는 속-종 레이블)의 존재를 특별히 고려하여 EOL, 통합 분류 정보 시스템(ITIS) [43] 및 iNaturalist의 분류 계층 구조를 신중하게 통합하고 백필합니다. 이 프로세스, 과제, 솔루션 및 남아 있는 문제에 대한 자세한 내용은 부록 C를 참조하십시오.

표 1. TREEOFLIFE-10M에 사용된 교육 데이터 소스. 소스 간에 분류 레이블을 통합하고 표준화합니다(§2.2).
2.3. 공개 및 통계
표 1은 데이터 세트 통계를 보여줍니다. TREEOFLIFE-10M은 450K개 이상의 고유한 분류명에 걸쳐 1,000만 개 이상의 이미지를 보유하고 있습니다. 그림 2는 문(phyla) 및 각각의 하위 분류군(주문부터 가족까지)별 이미지 분포를 보여줍니다.
선별된 교육 및 테스트 데이터 세트(TREEOFLIFE-10M 및 RARE SPECIES, §4.2에 설명됨)는 퍼블릭 도메인 포기 하에 Hugging Face(DOI 포함)에서 사용할 수 있으며, 기본 소스 라이선스가 허용하는 범위까지 가능합니다. 여기에는 이미지 메타데이터가 포함된 CSV와 기본 소스 링크, 데이터 세트를 생성하는 스크립트가 포함된 GitHub 리포지토리가 포함됩니다.4
3. 모델링
BIOCLIP은 OpenAI의 공개 CLIP 체크포인트에서 초기화되고 CLIP의 다중 모드 대조 학습 목표를 사용하여 TREEOFLIFE-10M에서 지속적으로 사전 학습됩니다.
3.1. 왜 CLIP인가?
일반 도메인 컴퓨터 비전 작업과 비교할 때 생물학 도메인의 가장 두드러진 차이점 중 하나는 풍부한 레이블 공간입니다. 분류군 레이블은 양이 많을 뿐만 아니라(2022년 기준 200만 종 이상 기록됨[44]) 계층적 분류 체계에서 서로 연결되어 있습니다. 이는 만족스러운 적용 범위와 일반화를 달성할 수 있는 우수한 기초 모델을 교육하는 데 어려움이 됩니다. 그럼에도 불구하고 수 세기 동안의 생물학 연구를 통해 축적된 레이블 공간의 복잡한 구조는 더 나은 일반화를 학습하기 위한 매우 풍부한 신호를 제공합니다. 직관적으로 레이블 공간의 구조가 기초 모델에 성공적으로 인코딩되면 모델이 특정 종을 본 적이 없더라도 해당 종의 해당 속 또는 과에 대한 좋은 표현을 학습했을 가능성이 높습니다. 이러한 계층적 표현은 새로운 분류군의 few-shot 또는 심지어 zero-shot 학습을 가능하게 하는 강력한 사전 역할을 합니다.
ResNet [33] 및 Swin Transformer [48]과 같은 많은 비전 기반 모델은 지도 분류 목표를 채택하고 입력 이미지에서 클래스 인덱스로의 매핑을 직접 학습합니다. 결과적으로 각 클래스 레이블은 별개의 기호로 취급되고 관계는 무시됩니다. 우리 작업의 핵심적인 깨달음은 CLIP에 사용된 다중 모드 대조 학습 목표를 레이블 공간의 계층적 구조를 활용하기 위해 용도를 변경할 수 있다는 것입니다. 이것은 명확한 선택이 아닙니다. 결국 TREEOFLIFE-10M은 주로 클래스 레이블로 레이블이 지정되며 이미지 캡션과 같은 자유 형식 텍스트로 레이블이 지정되지 않습니다. 자기 회귀 텍스트 인코더는 나중에 분류 순위 표현을 더 높은 순위에 조건화하여 분류 계층 구조를 자연스럽게 조밀한 레이블 공간에 포함합니다(그림 1). 계층적 분류[9, 11, 96]도 분류 체계를 활용할 수 있지만, CLIP 스타일의 대조 학습은 일반화를 크게 개선한다는 것을 경험적으로 보여줍니다(§4.4). 분류법에 부합하는 계층적 표현을 학습하기 위해 CLIP의 다중 모드 대조 학습 목표를 용도 변경하는 것은 새롭고 중요하지 않은 기술적 기여라는 점에 유의하십시오.
CLIP는 두 개의 단일 모드 임베딩 모델, 비전 인코더 및 텍스트 인코더를 훈련하여 (1) 긍정적인 (이미지, 텍스트) 쌍 간의 기능 유사성을 극대화하고 (2) 부정적인 (이미지, 텍스트) 쌍 간의 기능 유사성을 최소화합니다. 여기서 긍정적인 쌍은 훈련 데이터에서 가져오고 부정적인 쌍은 배치의 다른 모든 가능한 (이미지, 텍스트) 쌍입니다. 훈련 후 CLIP의 인코더 모델은 각 모드의 개별 인스턴스를 공유 기능 공간에 포함합니다. 다음으로 분류 구조를 통합하기 위해 CLIP에 대한 텍스트 입력 형식 지정에 대해 설명합니다.
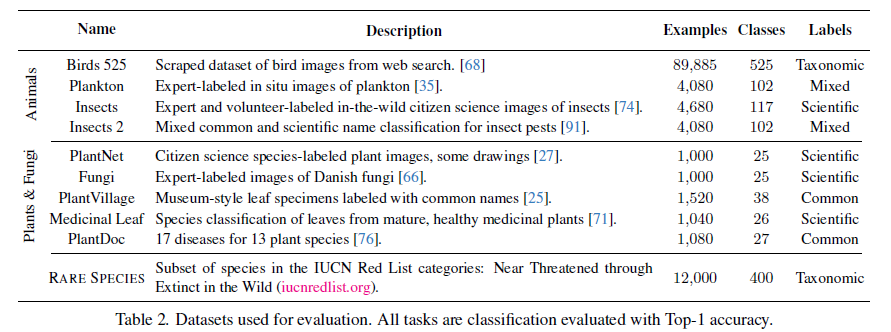
표 2. 평가에 사용된 데이터 세트. 모든 작업은 Top-1 정확도로 평가되는 분류입니다.

표 3. BIOCLIP 교육에서 고려되는 텍스트 유형.
3.2. 텍스트 유형
CLIP의 장점 중 하나는 텍스트 인코더가 자유 형식 텍스트를 허용한다는 것입니다. 생물학에서는 다른 분류 작업과 달리 클래스 이름의 형식이 다양합니다. 다음 사항을 고려합니다.
분류학적 이름: 더 높은 수준에서 더 낮은 수준까지 표준 7단계 생물학 분류는 kingdom, phylum, class, order, family, genus 및 species입니다. 각 종에 대해 루트에서 리프까지 모든 레이블을 단일 문자열로 연결하여 분류 체계를 "평평하게" 만듭니다. 이를 분류학적 이름이라고 합니다.
학명: 학명은 속과 종으로 구성됩니다(예: Pica hudsonia).
일반 이름: 분류 범주는 일반적으로 라틴어이며 일반 이미지 텍스트 사전 훈련 데이터 세트에서는 흔히 볼 수 없습니다. 대신 "black-billed magpie"와 같은 일반적인 이름이 더 널리 퍼져 있습니다. 일반 이름은 분류군에 1:1 매핑이 없을 수 있습니다. 단일 종에 여러 일반 이름이 있거나 동일한 일반 이름이 여러 종을 나타낼 수 있습니다.
BIOCLIP의 특정 다운스트림 사용 사례의 경우 학명과 같은 한 가지 유형의 레이블만 사용할 수 있는 경우가 있습니다. 추론 시간에 유연성을 높이기 위해 혼합 텍스트 유형 훈련 전략을 제안합니다. 각 훈련 단계에서 각 입력 이미지를 사용 가능한 모든 텍스트 유형에서 무작위로 샘플링된 텍스트와 쌍으로 만듭니다(표 3에 표시됨). 우리는 경험적으로 이 간단한 전략이 분류학적 이름의 일반화 이점을 유지하면서 추론 시간에 다른 이름을 사용할 때 더 많은 유연성을 제공한다는 것을 보여줍니다 (§4.3). CLIP에 대한 최종 텍스트 입력은 표준 CLIP 템플릿의 이름입니다(예: "Pica hudsonia 사진").
4. 실험
TREEOFLIFE-10M에 대해 BIOCLIP을 교육하고 BIOCLIP을 일반 비전 모델과 비교하고 모델링 선택이 BIOCLIP의 성능에 어떤 영향을 미치는지 조사합니다.
4.1. 훈련 및 평가 세부 정보
BIOCLIP을 훈련하기 위해 ViT-B/16 비전 트랜스포머[22] 이미지 인코더와 77 토큰 인과 관계 자기회귀 변환 텍스트 인코더를 사용하여 OpenAI의 CLIP 가중치[69]에서 초기화합니다. 코사인 학습률 스케줄[49]을 사용하여 TREEOFLIFE-10M에 대해 100 에포크 동안 사전 훈련을 계속합니다. 2개 노드에서 8개의 NVIDIA A100-80GB GPU를 사용하여 32,768개의 글로벌 배치 크기로 훈련합니다. 또한 iNat21 데이터 세트에 대해서만 기준선 모델을 훈련하고 TREEOFLIFE-10M(4.3절 및 4.4절)에서 무작위로 샘플링된 1M 예제에 대해 여러 절제 모델을 훈련하며, 1개 노드에서 4개의 NVIDIA A100 GPU에서 16,384개의 더 작은 글로벌 배치 크기를 제외하고는 BIOCLIP에 대해 동일한 절차를 따릅니다. 모든 하이퍼파라미터 및 교육 세부 정보는 부록 D에 있으며 교육 및 평가 코드는 공개적으로 사용할 수 있습니다.
Meta-Album[84], Birds 525[68] 및 새로운 RARE SPECIES 작업(§4.2에 설명됨)의 8가지 생물학적으로 관련된 작업인 10가지 분류 작업에 대해 평가합니다. Meta-Album은 다양한 주제를 포괄하는 메타 학습을 위한 데이터 세트 모음입니다. 구체적으로 Plankton, Insects, Insects 2, PlantNet, Fungi, PlantVillage, Medicinal Leaf 및 PlantDoc 데이터 세트를 사용합니다. 분류 작업은 생명 나무의 4개의 다세포 왕국(동물, 식물, 균류 및 원생생물)을 모두 포괄하며 다양한 이미지 분포(사진, 현미경 이미지, 그림 및 박물관 표본)를 가집니다. 표 2는 데이터 세트를 요약합니다. 전체 분류학적 이름부터 과학적 이름 또는 일반 이름만 포함하는 다양한 레이블 유형으로 구성됩니다.
Zero-shot 학습의 경우 CLIP와 동일한 절차를 따릅니다. Few-shot 학습의 경우 SimpleShot [90]을 따르고 가장 가까운 중심 분류기를 사용합니다. k-shot 학습의 경우 먼저 각 클래스에 대해 k개의 예제를 무작위로 샘플링하고 사전 훈련된 모델의 시각적 인코더에서 이미지 임베딩을 얻습니다. 그런 다음 k개의 임베딩의 평균 특징 벡터를 각 클래스의 중심으로 계산합니다. 데이터 세트에 남아 있는 모든 예제는 테스트에 사용됩니다. 각 중심 및 테스트 특징 벡터에 평균 빼기 및 L2 정규화를 적용한 후 테스트 벡터에 가장 가까운 중심을 갖는 클래스를 예측으로 선택합니다. 서로 다른 무작위 시드를 사용하여 각 few-shot 실험을 5회 반복하고 표 4에 평균 정확도를 보고합니다. 표준 편차가 있는 결과는 부록 E에 보고됩니다.
BIOCLIP을 원래 OpenAI CLIP [69] 및 LAION-400M [73]에서 훈련된 OpenCLIP [42]과 비교합니다. 직관적으로 유기체의 일반적인 이름은 CLIP 및 OpenCLIP의 훈련 데이터에서 가장 널리 퍼져 있으며 이러한 모델은 일반적인 이름과 함께 가장 잘 작동합니다. 이것은 또한 예비 테스트에서 확인됩니다. 따라서 데이터 세트에서 사용할 수 없는 경우 기본적으로 CLIP 및 OpenCLIP에 대한 클래스 레이블로 일반 이름을 사용합니다. BIOCLIP은 분류학적 이름을 활용할 수 있으므로 기본적으로 분류학적 + 일반 이름을 사용합니다. 그러나 표 2에서 볼 수 있듯이 테스트 데이터 세트에는 다양한 레이블이 있습니다. 선호하는 레이블 유형을 사용할 수 없는 경우 데이터 세트와 함께 제공되는 레이블을 사용합니다. 또한 few-shot 분류를 위해 ImageNet-21K [21] 사전 훈련 모델 [78] 및 DINO [15]와 비교합니다.
4.2. BIOCLIP은 보이지 않는 분류군으로 일반화할 수 있습니까?
생물학자들이 새로운 종과 기존 종을 발견하고 분류함에 따라 분류학적 이름이 추가, 제거 및 변경됩니다. BIOCLIP은 새로운 종마다 재훈련을 피하기 위해 보이지 않는 분류학적 이름으로 일반화해야 합니다. BIOCLIP이 보이지 않는 분류군에 잘 일반화되는지 여부를 경험적으로 답하기 위해 생물학적으로나 경험적으로 동기를 부여하는 새로운 평가 작업인 RARE SPECIES를 소개합니다.
"희귀" 종을 분류하는 것은 특히 지구 보존 노력[83]의 맥락에서 생물학에서 중요하고 어려운 컴퓨터 비전 응용 프로그램입니다. 우리가 아는 한, 다양하고 공개적으로 사용 가능한 희귀종 분류 데이터 세트는 없습니다. 최근에 발표된 연구[47, 56]는 12개의 클래스만 포함하여 종 다양성이 부족합니다. 우리는 이러한 격차를 해소하기 위해 IUCN 적색 목록(iucnredlist.org)에 있는 모든 ⇡ 25K 종을 수집하여 Near Threatened, Vulnerable, Endangered, Critically Endangered 또는 Extinct in the Wild로 분류합니다5. EOL 데이터 세트에서 최소 30개의 이미지로 표시되는 400개의 종을 선택한 다음 TREEOFLIFE-10M에서 제거하여 종당 30개의 이미지가 포함된 보이지 않는 RARE SPECIES 테스트 세트를 만듭니다. 이 데이터 세트는 (1) 보이지 않는 분류군에 대한 BIOCLIP의 분포 외 일반화, (2) BIOCLIP의 잠재적 응용 프로그램을 보여주고, (3) 커뮤니티가 지속적인 생물 다양성 위기를 해결하는 데 중요한 데이터 세트를 제공합니다.
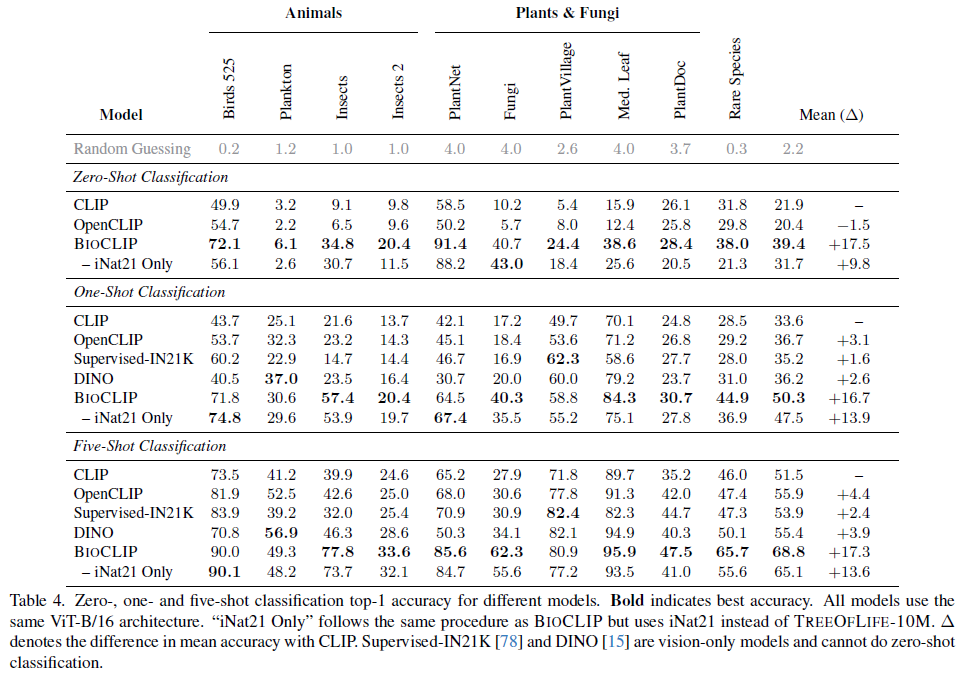
표 4. 서로 다른 모델에 대한 0, 1 및 5 shot 분류 상위 1 정확도. 굵게 표시된 것은 최고의 정확도를 나타냅니다. 모든 모델은 동일한 ViT-B/16 아키텍처를 사용합니다. "iNat21 Only"는 BIOCLIP과 동일한 절차를 따르지만 TREEOFLIFE-10M 대신 iNat21을 사용합니다.
결과. 표 4는 BIOCLIP이 특히 보이지 않는 분류군에서 zero-shot 분류에서 기준 CLIP 모델과 iNat21 훈련 CLIP 모델 모두를 훨씬 능가한다는 것을 보여줍니다("Rare Species" 열 참조). BIOCLIP의 광범위하고 다양한 작업 세트에 대한 강력한 zero-shot 성능은 TREEOFLIFE-10M의 광범위하고 다양한 클래스 때문입니다. §4.3에서 데이터 다양성이 광범위하게 유용한 이미지 표현으로 이어지는 방법을 살펴봅니다.
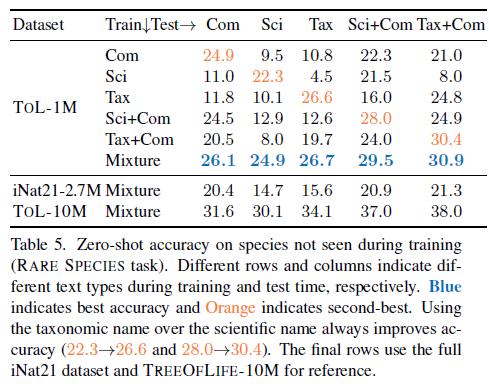
표 5. 훈련 중에 보이지 않는 종에 대한 제로샷 정확도 (RARE SPECIES 작업). 다른 행과 열은 각각 훈련 및 테스트 시간 동안 다른 텍스트 유형을 나타냅니다. 파란색은 최고의 정확도를 나타내고 주황색은 두 번째로 좋은 정확도를 나타냅니다. 학명보다 분류학적 이름을 사용하면 항상 정확도가 향상됩니다(22.3!26.6 및 28.0!30.4). 마지막 행은 참조용으로 전체 iNat21 데이터 세트 및 TREEOFLIFE-10M을 사용합니다.
4.3. 텍스트 유형이 일반화에 미치는 영향은 무엇입니까?
TREEOFLIFE-10M의 10% 하위 집합(계산 제약으로 인해 10%)에 대해 BIOCLIP을 교육하여 서로 다른 텍스트 유형이 zero-shot 일반화에 어떤 영향을 미치는지 조사합니다. 테스트 클래스에는 모든 텍스트 유형이 있고 모든 종이 교육에서 제외되어 보이지 않는 분류군에 대한 일반화를 테스트하는 데 이상적이므로 Rare Species 데이터 세트를 사용합니다. 이전 연구에서는 캡션의 다양성이 더 강력한 비전 모델을 만든다는 사실을 발견했으며[57], 훈련 중에 각 이미지에 대해 단일 고정 캡션[72]이 아닌 5가지 캡션 중 하나를 무작위로 사용합니다. 마찬가지로 혼합 텍스트 유형 전략(§3.2)을 사용합니다. 그것은 성능에 어떤 영향을 미칩니까?
결과. zero-shot 절제 결과는 표 5에 나와 있습니다. 몇 가지 눈에 띄는 관찰이 있습니다. 첫째, 분류학적 이름과 일반 이름을 함께 사용하면 가장 강력한 성능을 얻을 수 있으며, 일반화를 위해 분류 구조를 통합하는 것의 중요성을 보여줍니다. 둘째, 교육에 단일 텍스트 유형만 사용하는 경우 테스트 시 다른 텍스트 유형을 사용하면 성능이 크게 저하됩니다. 훈련에 혼합 텍스트 유형을 사용하면 테스트 중 모든 텍스트 유형에서 일관되게 강력한 성능을 얻을 수 있습니다. 이러한 결과는 혼합 텍스트 유형 사전 훈련이 분류학적 이름 사용의 일반화 이점을 크게 유지하면서 추론을 위한 다양한 텍스트 유형의 유연성도 제공함을 나타냅니다. 이는 다양한 다운스트림 작업에 사용될 수 있는 기본 모델의 중요한 속성입니다. 마지막으로 TREEOFLIFE-10M에서 100만 개의 예제를 사용하는 것은 iNat21에서 270만 개의 예제를 사용하는 것보다 성능이 뛰어나 TREEOFLIFE-10M에서 추가된 데이터 다양성의 중요성을 다시 한 번 확인시켜 줍니다.
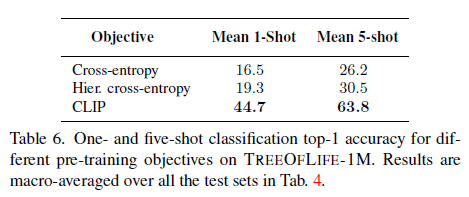
표 6. TREEOFLIFE-1M에 대한 다양한 사전 교육 목표에 대한 1샷 및 5샷 분류 상위 1 정확도. 결과는 표 4의 모든 테스트 세트에 대해 매크로 평균됩니다.
4.4. CLIP 목표가 필요한가요?
레이블이 지정된 이미지 데이터 세트에서 CLIP 목표를 사용하여 사전 훈련하는 것은 직관적이지 않은 결정입니다(Goyal et al. [29]는 CLIP 목표를 사용하여 미세 조정하지만 사전 훈련하지는 않음). 우리는 교차 엔트로피 분류 손실과 멀티 태스크 계층 변형을 사용하여 TREEOFLIFE-1M에 대해 두 개의 ViT-B/16 모델을 훈련함으로써 선택을 정당화한 다음 few-shot 설정에서 CLIP 목표와 비교합니다. 멀티 태스크 계층 훈련 목표는 분류 체계의 각 수준에 대해 교차 엔트로피를 사용하여 kingdom, phylum 등에서 종까지 레이블을 예측한 다음 해당 손실을 합산하는 것입니다[11]. 의사 코드는 목록 1에 제공됩니다.
결과. CLIP이 아닌 모델은 zero-shot 분류를 수행할 수 없기 때문에 1-shot 및 5-shot 설정에서만 동일한 10가지 작업 세트에서 각 모델을 평가합니다. 표 6에 평균 정확도를 보고합니다. 계층적 분류 모델은 단순 분류보다 성능이 뛰어나며 CLIP 기준선과 비슷합니다(표 4 참조). 그러나 CLIP 목표는 두 기준선 모두를 훨씬 능가하며 CLIP 목표의 용도 변경을 강력하게 정당화합니다.
4.5. BIOCLIP은 종 이상을 분류할 수 있습니까?
BIOCLIP은 (대조적인) 종 분류 목표에 대해 훈련되므로 종 분류를 넘어서는 사용이 제한될 수 있습니다. PlantVillage 및 PlantDoc 데이터 세트를 사용하여 식물 진단을 고려합니다. 여기서는 종과 질병(있는 경우)을 모두 분류해야 합니다. 대규모 데이터 레이블 지정은 비용이 많이 들지만 생물학자들은 항상 현장 가이드 또는 박물관 컬렉션에 대한 여러 인스턴스에 레이블을 지정합니다. 따라서 few-shot 분류는 이러한 종류의 작업 전송에 이상적인 설정입니다.
결과. BIOCLIP은 zero-shot 및 few-shot 설정(표 4의 PlantVillage 및 PlantDoc 참조) 모두에서 시각적 증상을 기반으로 식물 질병을 분류하는 데 있어 기준선을 능가합니다. Radford et al. [69]는 CLIP one-shot 및 two-shot 분류가 종종 zero-shot보다 성능이 좋지 않다는 것을 발견했습니다(few-shot 설정은 클래스 이름의 의미 정보를 사용할 수 없기 때문에). BIOCLIP은 레이블이 지정된 예제가 하나만 있어도 유용한 유용한 시각적 표현을 학습했습니다. BIOCLIP의 평균 1-shot 정확도는 zero-shot 정확도보다 9.1% 높습니다.
4.6. BIOCLIP은 계층 구조를 학습합니까?
BIOCLIP은 외부 평가에서 낮은 데이터 체제에서 강력한 성능을 보여주지만 그 이유는 무엇입니까? 우리는 또한 BIOCLIP의 학습된 이미지 표현을 시각화하여 이 질문에 대한 통찰력을 제공하기 위해 본질적인 평가를 수행합니다(그림 3). t-SNE[85]를 사용하여 iNat21의 검증 세트(훈련 중 보이지 않음)에서 이미지 표현을 포함하고 이미지의 분류 레이블별로 점을 색칠합니다. 각 플롯에 대해 레이블이 지정된 분류 순위 아래에 있는 예제 하위 집합에 대해 t-SNE를 독립적으로 실행합니다. 각 플롯은 하나의 분류 계층 순위와 다음 순위의 상위 6개 범주를 시각화합니다. 예를 들어 왼쪽 상단 플롯은 Animalia 왕국에서 가장 흔한 6개의 문을 시각화합니다. 왕국(공간상 생략) 및 문과 같은 상위 계급에서는 CLIP과 BIOCLIP 모두 분리가 잘 되지만 BIOCLIP의 표현은 더 세밀하고 더 풍부한 클러스터링 구조를 포함합니다. 하위 계급에서 BIOCLIP은 더 분리 가능한 특징을 생성하는 반면 CLIP의 특징은 복잡하고 명확한 구조가 부족합니다. 부록 F에는 더 많은 정성적 결과와 시각 자료가 있습니다.
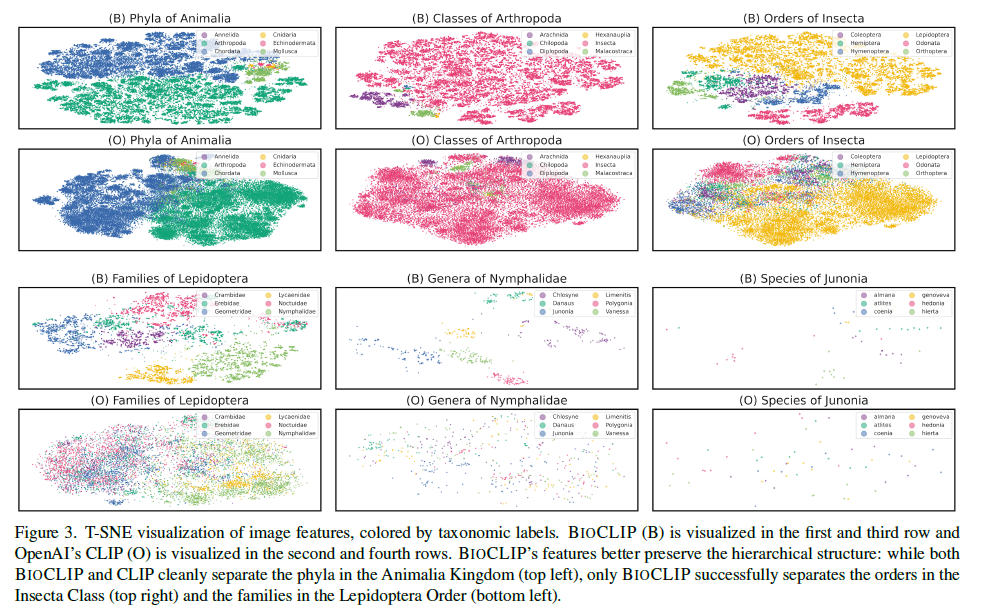
그림 3. 분류학적 레이블별로 색상이 지정된 이미지 특징의 T-SNE 시각화. BIOCLIP (B)은 첫 번째 및 세 번째 행에 시각화되고 OpenAI의 CLIP (O)는 두 번째 및 네 번째 행에 시각화됩니다. BIOCLIP의 기능은 계층적 구조를 더 잘 보존합니다. BIOCLIP과 CLIP 모두 Animalia Kingdom(왼쪽 상단)의 문을 깔끔하게 분리하지만 BIOCLIP만 Insecta Class(오른쪽 상단) 및 Lepidoptera Order(왼쪽 하단)의 주문을 성공적으로 분리합니다.
5. 관련 연구
다중 모드 기초 모델 훈련 데이터. CLIP [69]는 이미지 검색에 최적화된 대조 목표를 사용하여 노이즈가 많고 웹 규모(1억 개 이상)의 이미지-텍스트 데이터 세트에서 최첨단 비전 모델을 훈련했습니다. ALIGN [45] 및 BASIC [65]은 훈련 예제 수를 4억에서 66억으로 추가로 확장하여 비전 표현 품질을 개선했습니다. 그러나 추가 연구 [24, 26, 57, 93, 94]에서는 모두 데이터 세트 다양성과 이미지 및 캡션 의미 체계 간의 더 나은 정렬이 데이터 세트 크기보다 더 중요하며 다운스트림 작업에서 더 강력한 성능으로 이어진다는 것을 발견했습니다. TREEOFLIFE-10M은 iNat21의 10K에 440K 이상의 클래스를 추가하고 더 강력한 zero-shot 성능을 제공하여 다양성의 중요성을 강조합니다.
도메인별 CLIP. 도메인별 교육은 종종 일반 교육보다 뛰어나지만[18, 30], 주제 전문가는 종종 대규모 도메인별 데이터 세트에 레이블을 지정하기에는 너무 비쌉니다. 따라서 이미지-텍스트 훈련은 모델이 노이즈가 있는 이미지-텍스트 쌍에서 학습할 수 있기 때문에 특히 강력합니다. Ikezogwo et al. [41] 및 Lu et al. [50]은 계산 병리학을 위해 100만 개 이상의 이미지-텍스트 쌍을 수집했습니다. 우리는 클래스 다양성을 강조하면서 이미지의 10배를 수집합니다.
5. 관련 연구
컴퓨터 비전의 계층 구조. 컴퓨터 비전의 계층 구조는 ImageNet[70] 클래스가 계층적 WordNet[55]에서 가져온 것이기 때문에 잘 연구되어 있습니다. Bilal et al.[10]은 ImageNet에 대한 모델 예측을 연구하고 모델 혼동 패턴이 계층적 클래스 구조를 따른다는 것을 발견했습니다. 계층 구조를 AlexNet의 아키텍처[46]에 통합하고 ImageNet top-1 오류를 8% 절대적으로 개선했습니다. Bertinetto et al.[9]은 이미지 분류기의 실수 심각도를 측정하고 계층 구조를 통합하는 대체 목표를 제안하여 상위 1 정확도를 악화시키는 대신 실수 심각도를 줄입니다. Zhang et al.[96]은 레이블 간의 계층적 거리가 임베딩 공간에서 원하는 거리에 해당하는 대조 목표를 제안하고 ImageNet 및 iNat17[88]에서 교차 엔트로피보다 성능이 뛰어납니다. 우리는 용도 변경된 CLIP 목표를 통해 454K 고유 클래스에 계층적 분류를 적용하는 반면 이전 연구에서는 더 작은 레이블 공간에 계층 구조를 적용했습니다.
생물학을 위한 컴퓨터 비전. 세분화된 분류는 컴퓨터 비전의 고전적인 과제이며 생물학적 이미지는 종종 모델 벤치마킹에 사용됩니다. Berg et al.[8], Piosenka[68], Wah et al.[89]은 모두 조류 종 분류를 사용하여 세분화된 분류 능력을 평가합니다. 생물학 작업은 대조 학습 프레임워크[20, 92], 약하게 감독되는 객체 감지[19] 및 반지도 학습 방법[34]에 사용됩니다.
6. 결론
대규모의 다양한 생물 이미지 데이터 세트인 TREEOFLIFE-10M과 생명의 나무를 위한 기초 모델인 BIOCLIP을 각각 소개합니다. 광범위한 평가를 통해 BIOCLIP은 zero-shot 및 few-shot 설정 모두에서 생물학을 위한 강력한 세분화된 분류기임을 보여줍니다. 보이지 않는 종에 대한 절제를 통해 전체 분류학적 이름을 사용하는 것이 다른 캡션 유형보다 더 강력한 일반화를 가져온다는 가설을 확인하고 BIOCLIP 표현을 시각화하여 BIOCLIP 임베디드 이미지가 분류 계층 구조와 더 잘 일치한다는 것을 발견했습니다.
CLIP 목표는 450K개 이상의 분류군에 대해 시각적 표현을 효율적으로 학습하지만 BIOCLIP은 기본적으로 분류를 수행하도록 훈련됩니다. 향후 작업에서는 iNaturalist에서 1억 개 이상의 research-grade 이미지를 통합하는 등 데이터 규모를 더욱 확장하고 BIOCLIP이 세분화된 특성 수준 표현을 추출할 수 있도록 종의 모양에 대한 더 풍부한 텍스트 설명을 수집할 것입니다.
감사의 말
귀중한 피드백을 제공해 주신 Imageomics 팀(ArXiv에 전체 팀 목록이 있음), 데이터를 공유해 주신 BIOSCAN-1M 및 iNaturalist 팀, EOL 이미지 액세스를 도와주신 EOL의 Jennifer Hammock에게 감사드립니다. 우리의 연구는 NSF OAC 2118240 및 Ohio Supercomputer Center [16]의 리소스 지원을 받습니다.
-----------------------------------------------------------------------------------------------------------------------------------------------------------------
- 분류 체계의 계층 구조 활용: BIOCLIP은 텍스트 인코딩 과정에서 생물학 분류 체계의 계층 구조를 반영하여 종 간의 유사성을 파악하고 일반화 능력을 향상시켰습니다. 이는 단순히 데이터셋의 양적인 측면뿐만 아니라 질적인 측면에서도 모델의 성능 향상에 기여했습니다.
- 다양한 텍스트 유형 활용: BIOCLIP은 훈련 과정에서 다양한 텍스트 유형을 혼합하여 사용함으로써 특정 텍스트 유형에 국한되지 않고 다양한 방식으로 종을 인식하고 분류하는 능력을 갖추게 되었습니다.
-----------------------------------------------------------------------------------------------------------------------------------------------------------------
'인공지능' 카테고리의 다른 글
| SegFormer: Simple and Efficient Design for Semantic Segmentation with Transformers (2) | 2024.07.10 |
|---|---|
| Better & Faster Large Language Models via Multi-token Prediction (3) | 2024.07.09 |
| Mip-Splatting: Alias-free 3D Gaussian Splatting (1) | 2024.07.07 |
| Rich Human Feedback for Text-to-Image Generation (1) | 2024.07.06 |
| Generative Image Dynamics (1) | 2024.07.05 |



