https://arxiv.org/abs/1606.06650
3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation
This paper introduces a network for volumetric segmentation that learns from sparsely annotated volumetric images. We outline two attractive use cases of this method: (1) In a semi-automated setup, the user annotates some slices in the volume to be segment
arxiv.org
초록
이 논문은 희소하게 주석된 볼륨 이미지를 학습하는 볼륨 세분화를 위한 네트워크를 소개합니다. 이 방법의 두 가지 매력적인 사용 사례를 설명합니다: (1) 반자동 설정에서는 사용자가 세분화될 볼륨의 일부 슬라이스에 주석을 달고, 네트워크는 이러한 희소한 주석에서 학습하여 조밀한 3D 세분화를 제공합니다. (2) 완전 자동화된 설정에서는 대표적인 희소 주석 학습 세트가 존재한다고 가정합니다. 이 데이터 세트로 학습된 네트워크는 새로운 볼륨 이미지를 조밀하게 세분화합니다. 제안된 네트워크는 Ronneberger 등에서 제안한 이전 u-net 아키텍처를 확장하여 모든 2D 연산을 3D 연산으로 대체합니다. 이 구현은 훈련 중 효율적인 데이터 증강을 위해 실시간 탄성 변형을 수행합니다. 네트워크는 처음부터 끝까지 엔드-투-엔드로 학습되며, 사전 학습된 네트워크는 필요하지 않습니다. 우리는 복잡하고 변동성이 큰 3D 구조인 Xenopus 신장을 대상으로 제안된 방법의 성능을 테스트했으며, 두 가지 사용 사례 모두에서 좋은 결과를 얻었습니다.
키워드: 컨볼루션 신경망, 3D, 생의학 볼륨 이미지 세분화, Xenopus 신장, 반자동, 완전 자동, 희소 주석
1. 서론

그림 1: 3D u-net을 이용한 볼륨 세분화의 응용 시나리오. (a) 반자동 세분화: 사용자가 세분화할 각 볼륨의 일부 슬라이스에 주석을 달면, 네트워크가 조밀한 세분화를 예측함. (b) 완전 자동 세분화: 네트워크는 대표적인 학습 세트에서 주석이 달린 슬라이스로 학습된 후, 주석이 없는 볼륨에서도 실행될 수 있음.
생의학 데이터 분석에서는 볼륨 데이터가 풍부합니다. 이러한 데이터를 세분화 레이블로 주석을 달면 어려움이 발생하는데, 컴퓨터 화면에서는 2D 슬라이스만 표시할 수 있기 때문입니다. 따라서 슬라이스 단위로 대형 볼륨을 주석 다는 작업은 매우 번거롭습니다. 이 작업은 비효율적이기도 합니다. 왜냐하면 인접한 슬라이스들은 거의 동일한 정보를 나타내기 때문입니다. 특히 학습 기반 접근법에서는 상당한 양의 주석이 달린 데이터가 필요한데, 3D 볼륨을 완전히 주석하는 것은 잘 일반화할 수 있는 대규모 및 풍부한 학습 데이터 세트를 생성하는 효과적인 방법이 아닙니다.
이 논문에서는 조밀한 볼륨 세분화를 생성하는 법을 학습하는 딥 네트워크를 제안하지만, 학습에는 몇 개의 주석이 달린 2D 슬라이스만 필요합니다. 이 네트워크는 그림 1에 묘사된 두 가지 방식으로 사용할 수 있습니다: 첫 번째 응용 사례는 희소하게 주석이 달린 데이터 세트를 조밀하게 만드는 것이 목표이며, 두 번째는 다수의 희소하게 주석이 달린 데이터 세트로부터 학습하여 새로운 데이터에 일반화하는 것입니다. 두 경우 모두 매우 중요합니다.
이 네트워크는 이전의 u-net 아키텍처를 기반으로 하며, 이미지 전체를 분석하기 위한 수축 인코더 부분과 전체 해상도의 세분화를 생성하는 연속적인 확장 디코더 부분으로 구성됩니다 [11]. u-net은 완전히 2D 아키텍처이지만, 이 논문에서 제안된 네트워크는 3D 볼륨을 입력으로 받아서 이를 처리하는 3D 연산, 특히 3D 컨볼루션, 3D 최대 풀링, 3D 업-컨볼루션 레이어를 사용합니다. 또한 네트워크 아키텍처에서 병목 현상을 피하고 [13], 빠른 수렴을 위해 배치 정규화 [4]를 사용합니다.
많은 생의학 응용에서, 네트워크가 합리적으로 잘 일반화할 수 있도록 학습시키기 위해서는 매우 적은 수의 이미지만 필요합니다. 이는 각각의 이미지가 이미 반복적인 구조와 그에 상응하는 변화를 포함하고 있기 때문입니다. 볼륨 이미지에서는 이 효과가 더욱 두드러져, 단 두 개의 볼륨 이미지로 네트워크를 학습시켜 세 번째 이미지에 일반화할 수 있습니다. 가중 손실 함수와 특별한 데이터 증강 방법을 통해, 네트워크는 수작업으로 주석된 소수의 슬라이스만으로, 즉 희소하게 주석된 학습 데이터로 학습할 수 있습니다.
우리는 제안된 방법이 Xenopus 신장의 어려운 공초점 현미경 데이터 세트에 성공적으로 적용된 사례를 보여줍니다. Xenopus 신장은 발달 과정에서 복잡한 구조를 형성하기 때문에 [7], 미리 정의된 매개변수 모델의 적용이 제한됩니다. 먼저 소수의 주석이 달린 슬라이스에서 조밀한 세분화를 만드는 질적 결과를 제공하여 그 품질을 입증합니다. 이러한 결과는 정량적 평가로 뒷받침됩니다. 또한 주석이 달린 슬라이스의 수가 네트워크 성능에 미치는 영향을 보여주는 실험도 제공합니다. Caffe[5] 기반의 네트워크 구현은 오픈 소스로 제공됩니다. (1 오픈소스 링크).
1.1 관련 연구
오늘날 어려운 생의학 2D 이미지는 CNN(Convolutional Neural Networks)을 통해 인간의 성능에 가까운 정확도로 세분화할 수 있습니다 [11, 12, 3]. 이러한 성공 덕분에, 생의학 볼륨 데이터에 3D CNN을 적용하려는 여러 시도가 이루어졌습니다. Milletari 등 [9]은 3D 세분화를 위해 Hough 투표 방법과 결합된 CNN을 제안했습니다. 그러나 그들의 방법은 엔드-투-엔드(end-to-end)가 아니며, 오직 콤팩트한 덩어리 형태의 구조에만 작동합니다. Kleesiek 등 [6]의 접근법은 3D 세분화를 위한 몇 안 되는 엔드-투-엔드 3D CNN 접근법 중 하나입니다. 그러나 그들의 네트워크는 깊지 않으며, 첫 번째 컨볼루션 후에 하나의 최대 풀링(max-pooling)만을 가지고 있어, 여러 스케일에서 구조를 분석할 수 없습니다.
우리의 연구는 2015년 여러 국제 세분화 및 추적 대회에서 우승한 2D u-net [11]을 기반으로 합니다. u-net의 아키텍처와 데이터 증강 기법은 소수의 주석이 달린 샘플로부터 매우 좋은 일반화 성능을 가진 모델을 학습할 수 있도록 해줍니다. 이는 올바르게 적용된 강체 변환과 약간의 탄성 변형이 생물학적으로 타당한 이미지를 여전히 생성할 수 있다는 사실을 활용합니다. 의미론적 세분화를 위한 완전 컨볼루션 네트워크 [8]와 u-net 같은 업-컨볼루션(up-convolutional) 아키텍처는 아직 널리 보급되지 않았으며, 그러한 아키텍처를 3D로 일반화하려는 시도는 Tran 등 [14]의 연구 하나만 존재합니다. Tran 등의 연구에서는 이 아키텍처가 비디오에 적용되었으며, 학습을 위한 완전한 주석이 제공되었습니다.
본 논문의 핵심은 희소하게 주석된 볼륨에서 처음부터 학습할 수 있으며, 매끄러운 타일링 전략을 통해 임의의 크기의 볼륨에서도 작동할 수 있다는 점입니다.
2. 네트워크 아키텍처

그림 2: 3D u-net 아키텍처. 파란색 상자는 특징 맵을 나타내며, 각 특징 맵 위에 채널 수가 표시됩니다.
그림 2는 네트워크 아키텍처를 보여줍니다. 표준 u-net과 마찬가지로, 네트워크는 네 개의 해상도 단계를 가진 분석 경로와 합성 경로를 각각 가지고 있습니다. 분석 경로에서, 각 레이어는 두 개의 3×3×3 컨볼루션을 포함하며, 각각은 정류된 선형 유닛(ReLu) 뒤에 위치하며, 그 후 각 차원에서 두 개씩의 스트라이드를 가지는 2×2×2 최대 풀링(max pooling)이 뒤따릅니다. 합성 경로에서는, 각 레이어가 각 차원에서 두 개씩의 스트라이드를 가지는 2×2×2 업컨볼루션으로 시작하며, 그 후에 두 개의 3×3×3 컨볼루션과 각각 ReLu가 뒤따릅니다. 분석 경로에서 동일한 해상도의 레이어에서 가져온 바로가기 연결(Shortcut connections)은 합성 경로에 필수적인 고해상도 특징을 제공합니다. 마지막 레이어에서는 1×1×1 컨볼루션을 사용하여 출력 채널 수를 라벨 수로 줄이는데, 우리 경우에는 3개의 라벨이 있습니다. 이 아키텍처는 총 19,069,955개의 파라미터를 가지고 있습니다. [13]에서 제안된 것처럼, 우리는 최대 풀링 이전에 채널 수를 두 배로 늘려 병목 현상을 피합니다. 이 방법은 합성 경로에서도 적용됩니다.
네트워크의 입력은 3개의 채널을 가진 132×132×116의 보셀 타일입니다. 최종 레이어에서 출력은 x, y, z 방향 각각에서 44×44×28 보셀이 됩니다. 보셀 크기가 1.76×1.76×2.04 μm³일 때, 예측된 세분화에서 각 보셀의 수용 영역은 대략 155×155×180 μm³가 됩니다. 따라서 각 출력 보셀은 효율적으로 학습할 수 있는 충분한 문맥을 가지고 있습니다.
우리는 또한 각 ReLU 전에 배치 정규화("BN")를 도입합니다. [4]에 따르면, 각 배치는 학습 중에 해당 배치의 평균과 표준 편차로 정규화되며, 이 값을 사용하여 전역 통계가 업데이트됩니다. 이후에는 명시적으로 스케일과 바이어스를 학습하는 레이어가 뒤따릅니다. 테스트 시에는 이 계산된 전역 통계와 학습된 스케일 및 바이어스를 통해 정규화가 이루어집니다. 그러나 우리는 배치 크기가 하나이고 샘플이 적습니다. 이러한 응용에서는 테스트 시에도 현재 통계를 사용하는 것이 가장 효과적입니다.
희소한 주석으로 학습할 수 있도록 해주는 아키텍처의 중요한 부분은 가중 소프트맥스 손실 함수입니다. 주석이 달리지 않은 픽셀의 가중치를 0으로 설정하면, 주석이 달린 픽셀만을 학습하고, 따라서 전체 볼륨에 일반화할 수 있습니다.
3. 구현 세부사항
3.1 데이터
우리는 Nieuwkoop-Faber 단계 36-37에 해당하는 Xenopus 신장 배아의 세 개의 샘플을 가지고 있습니다 [10]. 그 중 하나는 그림 1(왼쪽)에 나와 있습니다. 3D 데이터는 Zeiss LSM 510 DUO 반전 공초점 현미경을 사용하여 40배/1.3 오일 침지 대물렌즈가 장착된 Plan-Apochromat을 이용해 보셀 크기 0.88×0.88×1.02 μm³에서 세 개의 채널로 네 개의 타일로 기록되었습니다. 우리는 XuvTools [1]를 사용하여 타일을 큰 볼륨으로 스티칭했습니다. 첫 번째 채널은 488nm의 여기 파장에서 Tomato-Lectin이 Fluorescein과 결합된 것을 보여줍니다. 두 번째 채널은 405nm의 여기에서 DAPI로 염색된 세포 핵을 보여줍니다. 세 번째 채널은 564nm의 여기에서 Cy3로 표지된 2차 항체를 사용하여 세포막을 표시하는 Beta-Catenin을 보여줍니다. 우리는 Slicer3D [2]를 사용하여 각 볼륨에서 일부 직교하는 xy, xz 및 yz 슬라이스를 수작업으로 주석 처리했습니다. 주석 위치는 데이터의 좋은 대표성을 기준으로 선택되었으며, 즉 주석 슬라이스는 모든 3차원에서 가능한 한 균일하게 샘플링되었습니다. 다양한 구조물에는 0: "관 내", 1: "관", 2: "배경", 3: "주석 없음"이라는 레이블이 부여되었습니다. 주석이 달리지 않은 모든 보셀도 레이블 3("주석 없음")을 받습니다. 모든 실험은 각 차원에서 원래 해상도의 2배로 다운샘플링된 버전에서 수행되었습니다. 따라서 실험에 사용된 데이터 크기는 각각 샘플 1, 2, 3의 x×y×z 차원에서 248×244×64, 245×244×56 및 246×244×59입니다. 직교하는 (yz, xz, xy) 슬라이스에서 수동으로 주석 처리된 슬라이스의 수는 샘플 1, 2, 3에 대해 각각 (7, 5, 21), (6, 7, 12) 및 (4, 5, 10)입니다.
3.2 학습
회전, 스케일링 및 그레이 값 증강 외에도, 우리는 데이터와 실제 레이블 모두에 부드러운 조밀 변형 필드를 적용합니다. 이를 위해, 각 방향에서 32 보셀 간격의 그리드에서 표준 편차 4의 정규 분포로부터 임의의 벡터를 샘플링한 후, B-스플라인 보간법을 적용합니다. 네트워크 출력과 실제 레이블은 가중 크로스 엔트로피 손실이 적용된 소프트맥스를 사용하여 비교됩니다. 여기서 자주 나타나는 배경의 가중치는 줄이고, 내부 관의 가중치는 높여서 관과 배경 보셀이 손실에 미치는 영향을 균형 있게 조정합니다. 레이블 3("주석 없음")이 부여된 보셀은 손실 계산에 기여하지 않으며, 즉 가중치가 0입니다. 네트워크 학습을 위해 우리는 Caffe [5] 프레임워크의 확률적 경사 하강법(SGD) 솔버를 사용합니다. 큰 3D 네트워크 학습을 가능하게 하기 위해 메모리 효율적인 cuDNN [3] 컨볼루션 레이어 구현을 사용했습니다. 데이터 증강은 학습 중 실시간으로 이루어져, 학습 반복 횟수만큼 서로 다른 이미지가 생성됩니다. 우리는 NVIDIA TitanX GPU에서 약 3일 동안 약 70,000번의 학습 반복을 수행했습니다.
4. 실험
4.1 반자동 세분화
반자동 세분화의 경우, 사용자가 소수의 볼륨 이미지를 완전히 세분화해야 하며, 이전에 세분화된 데이터가 없다고 가정합니다. 제안된 네트워크는 사용자가 각 볼륨에서 몇 개의 슬라이스에 주석을 달고, 네트워크가 조밀한 볼륨 세분화를 생성할 수 있도록 해줍니다.
정성적 평가를 위해, 우리는 네트워크를 세 개의 희소하게 주석된 샘플 모두에서 학습시켰습니다. 그림 3은 우리의 세 번째 샘플에 대한 세분화 결과를 보여줍니다. 네트워크는 소수의 주석이 달린 슬라이스로부터 전체 3D 볼륨 세분화를 찾아내어, 전문가들이 전체 볼륨을 주석하는 작업을 대신해 줍니다.

그림 3: (a) 세 번째 Xenopus 신장의 공초점 촬영 이미지. (b) 배치 정규화가 적용된 제안된 3D u-net에 의한 결과적인 조밀한 세분화.
반자동 설정에서 정량적 성능을 평가하기 위해, 세 개의 샘플에서 수작업으로 주석 처리된 총 77개의 슬라이스를 균일하게 세 부분으로 나누고, 배치 정규화를 적용한 경우와 그렇지 않은 경우 모두에서 3겹 교차 검증(3-fold cross validation)을 수행했습니다. 이를 위해 테스트 슬라이스를 제거하고, 이들을 주석 처리되지 않은 상태로 두었습니다. 이는 사용자가 더 희소한 주석을 제공하는 상황을 시뮬레이션합니다. 전체 3D 컨텍스트를 사용하는 것의 이점을 측정하기 위해, 모든 주석된 슬라이스를 독립적인 이미지로 처리하는 순수 2D 구현 결과와 비교했습니다. 실험 결과는 표 2에 제시되어 있습니다. 정확도를 측정하기 위해 교차 엔트로피 손실과 비교하여, 예측된 3D 볼륨과 실제 값 간의 교차 비율(Intersection over Union, IoU)을 사용했습니다. IoU는 참 긍정/(참 긍정 + 거짓 부정 + 거짓 긍정)으로 정의됩니다. 결과는 제안된 접근 방식이 매우 적은 수의 주석된 슬라이스에서 이미 일반화할 수 있으며, 적은 주석 작업으로도 매우 정확한 3D 세분화를 이끌어낼 수 있음을 보여줍니다.
우리는 또한 주석된 슬라이스 수가 네트워크 성능에 미치는 영향을 분석했습니다. 이를 위해, 하나의 샘플에 대한 반자동 세분화를 시뮬레이션했습니다. 각 직교 방향에서 1개의 주석된 슬라이스를 사용하여 시작한 후, 주석된 슬라이스 수를 점진적으로 증가시켰습니다. 표 2에서 각 샘플(S1, S2, S3)에 대해 몇 개의 추가된 실제 값(“GT”) 슬라이스마다 네트워크의 높은 성능 향상을 보고합니다. 결과는 배치 정규화를 적용하여 10시간 동안 학습된 네트워크에서 얻은 것입니다. 테스트에는 이 실험의 어떤 설정에서도 사용되지 않은 슬라이스를 사용했습니다.
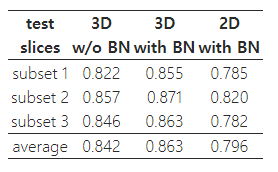
표 1: 반자동 세분화를 위한 교차 검증 결과 (IoU)
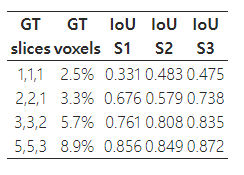
표 2: 반자동 세분화를 위한 슬라이스 수의 영향 (IoU)
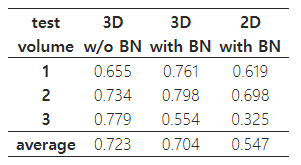
표 3: 완전 자동 세분화를 위한 교차 검증 결과 (IoU)
4.2 완전 자동 세분화
완전 자동 세분화 설정은 사용자가 비교 가능한 환경에서 기록된 다수의 이미지를 세분화하려고 한다고 가정합니다. 우리는 또한 대표적인 학습 데이터 세트를 구성할 수 있다고 가정합니다.
이 설정에서 성능을 추정하기 위해 두 개의 (부분적으로 주석이 달린) 신장 볼륨으로 학습하고, 학습된 네트워크를 사용하여 세 번째 볼륨을 세분화했습니다. 우리는 모든 가능한 학습 및 테스트 볼륨 조합에 대한 결과를 보고합니다. 표 3은 이전 섹션과 같이 제외된 볼륨의 모든 주석된 2D 슬라이스에 대한 IoU를 요약합니다. 이 실험에서 배치 정규화(BN)는 결과를 개선했으나, 세 번째 설정에서는 역효과를 보였습니다. 우리는 데이터 세트 간의 큰 차이가 이러한 효과를 초래한 것이라고 생각합니다. 완전 자동 세분화의 일반적인 사용 사례는 훨씬 더 큰 샘플 크기에서 작동하며, 동일한 수의 희소한 레이블을 훨씬 더 많은 데이터 세트에 쉽게 분배하여 더 대표적인 학습 데이터 세트를 얻을 수 있을 것입니다.
5. 결론
우리는 희소한 주석에서 3D 볼륨을 반자동 및 완전 자동으로 세분화하는 엔드-투-엔드 학습 방법을 소개했습니다. 이 방법은 Xenopus 신장의 고도로 변동하는 구조에 대해 정확한 세분화를 제공합니다. 반자동 설정에서 3겹 교차 검증 실험을 통해 평균 IoU 0.863을 달성했습니다. 완전 자동 설정에서는 3D 아키텍처가 동등한 2D 구현에 비해 성능 향상을 보여줍니다. 이 네트워크는 처음부터 학습되었으며, 이 응용을 위해 특별히 최적화되지 않았습니다. 우리는 이 방법이 다른 많은 생의학 볼륨 세분화 작업에도 적용될 수 있을 것으로 기대합니다. 이 구현은 오픈 소스로 제공됩니다.
감사의 말
이 연구를 지원해준 DFG(EXC 294 및 CRC-1140 KIDGEM 프로젝트 Z02 및 B07)에 감사를 드립니다. Ahmed Abdulkadir는 ZIM(Zentrales Innovationsprogramm Mittelstand)의 보조금 KF3223201LW3로부터 자금을 지원받았음을 밝힙니다. Soeren S. Lienkamp는 DFG(Emmy Noether-Programm)로부터 자금을 지원받았음을 밝힙니다. 또한 유용한 주석을 제공해준 Elitsa Goykovka와 우수한 이미징 기술 지원을 제공해준 Alena Sammarco에게도 감사드립니다.



